目次
- ( 前回の記事 )
- 1. 人類は、塩基を文字にもつ「ゲノム言語」をどこまで、操れるようになったのか?
- (MIT発)BioBrick: 機能単位の塩基配列ブロックを、積み木細工のように自在に組み合わせるアプローチ
- 2.人工知能・機械学習技術は、遺伝子工学・合成生物学の技術発展にどう関わっているのか?
- 次回の記事で論じること
( 前回の記事 )
前回の記事では、カーツワイル氏が21世紀の科学技術の基軸となる3つの技術として、Genome science(遺伝子学)・Nano-technology(ナノ・テクノロジー)・Robotics(ロボット工学+人工知能技術)に光を当てているものの、A.I.(人工知能)技術の進展が、これら3つの技術の発展に対して、どのような影響を与えているのかが主題として取り上げられていない事実を指摘しました。
Genome science(遺伝子学)は、人間が、遺伝子という言語を理解し、その遺伝子の構成(配列順序)を(人間が)書き換えることで、人間によって有用な生物試料(アミノ酸構成物)を創出する技術です。
同様に、Nano-technology(ナノ・テクノロジー)も、原子・分子スケールの力学法則に対する知見をもとに、人間がミクロ・スケールでの物質の配列構造を書き換え・再編成することで、人間によって有用な物質資源を創出する技術です。
この意味で、Genome science(遺伝子学)とNano-technology(ナノ・テクノロジー)はいずれも、(自然界が生み出した)なんらかの法則性・ルール(規則)を備えた「言語」が、どのような文法(言語規則)を宿した記号体系であるのかを(人類が)数理科学の技法を駆使して解読・理解する技術であると指摘することができます。
さらにこの両技術は、人間が(その)解読・理解した言語規則(文法)にのっとった(これまで自然界には存在しなかった)新しい「文章」を書くことで、(これまで自然界には存在しなかった)新しい塩基配列を持つ生物を生み出したり、(これまで自然界には存在しなかった)新しい分子構造の物質を生み出すことに道を開く技術であるという点でも、その本質を共有します。
その意味で、言語解読と文章構築に取り組む(人工知能技術における)自然言語処理技術(NLP: Natural Language Processing)は、Genome science(遺伝子学)とNano-technology(ナノ・テクノロジー)の技術発展に、なんらかの影響を与えているのではないか、という視点を、前回の記事で提示しました。
今回は、前回の記事に続く「カーツワイルのGNR論」の第2段として、以下の2つを主題として、カーツワイル氏の『ポスト・ヒューマン誕生』が刊行されて以後の技術の進展状況を論じていきます。
- G(遺伝学・ゲノム工学)はいまどこまで、A(アデニン)・T(チミン)・G(グアニン)・C(シトシン)を基本「文字」とする言語解読・文章構築技術として、成立してきているのか?
- A.I.(人工知能)技術の進展が、GNRのひとつであるGenome science(遺伝子学)の技術革新に対して、どのよう影響を与えているのか
(以下、Wikipedia日本語版「遺伝子」所収の遺伝子のイメージ図を転載)
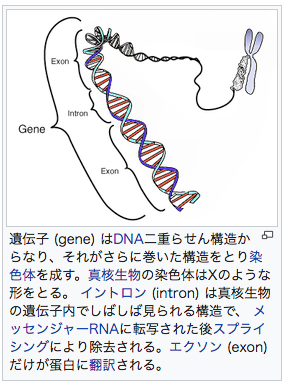
Wikipedia日本語版「遺伝子」所収の遺伝子のイメージ図を転載。
DNAの4つの塩基(アデニン・グアニン・シトシン・チミン)
地球上の生命体のからだは、20種類のアミノ酸が一次元方向に並んだ配列が、さまざまな立体構造に折り畳まれたタンパク質(人間の場合、およそ10万種類あるとされる)によって、構築されています。
生体内のタンパク質には、どのようなものがあるのかは、一例として以下を参照ください。
タンパク質は、アミノ酸の一次元配列が、立体構造に組み上げられたものです。この「立体構造」は、「タンパク質構造」と呼ばれています。
生化学的には、タンパク質の構造には4つの階層がある。
- 一次構造 – ペプチド鎖のアミノ酸の配列
- 二次構造 – 局所的に見られる、対称的な副構造で、1つのタンパク質分子の中に多くの種類の二次構造が含まれる
- 三次構造 – 1つの分子の三次元構造
- 四次構造 – いくつかのポリペプチドやタンパク質サブユニットの複合体
これらの構造の階層がある他に、タンパク質は機能の発現の過程で、構造が変化することがある。構造変化前後の三次構造や四次構造は異性体の関係にある。
上記のWikipedia「タンパク質構造」から、タンパク質の1次構造から4次構造までの模式図を転載します。
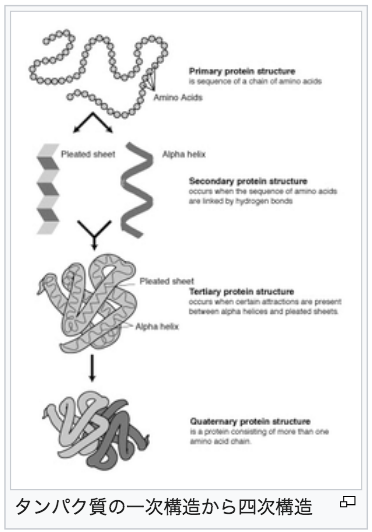
Wikipedia「タンパク質構造」より、タンパク質の1次構造から4次構造までの模式図を転載。
このうち、おおもととなるタンパク質の「一次構造」とは、「アミノ酸が一次元方向に並んだ配列」です。そして、どのアミノ酸とどのアミノ酸をどのような並び順序で(一次元に)並べていくのかを指定しているのが、DNAのなかにある「塩基」です。
「塩基」には、アデニン・グアニン・シトシン・チミンの4つの種類があります。
Wikipedia(英語版)から、それぞれの「塩基」の化学構造式を転載します。
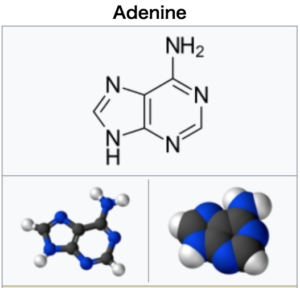 Wikipedia “Adenine”より転載。 https://en.wikipedia.org/wiki/Adenine |
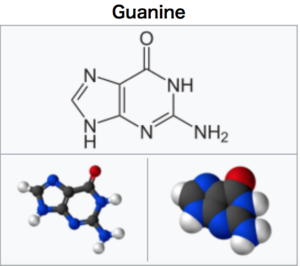 Wikipedia “Guanine”より転載。 https://en.wikipedia.org/wiki/Guanine |
 Wikipedia “Cytosine”より転載。 https://en.wikipedia.org/wiki/Cytosine |
 Wikipedia “Thymine”より転載。 https://en.wikipedia.org/wiki/Thymine |
DNA(デオキシリボ核酸)は、「脱酵素リボース」(デオキシリボース)と「リン酸」と「塩基(アデニン、グアニン、シトシン、チミンのうちどれか1つ)」の3者が化学結合した「dヌクレオシド一リン酸(dヌクレオチド,デオキシリボヌクレオチド」が、二重らせん構造で数珠つなぎになったものです。
どの「塩基」を含むのかによって、「dヌクレオチド」は、次の4つの異なる名称で呼ばれています。
- アデニル酸(アデノシン一リン酸、AMP)
- グアニル酸(グアノシン一リン酸、GMP)
- シチジル酸(シチジン一リン酸、CMP)
- ウリジル酸(ウリジン一リン酸、UMP)
Wikipedia(日本語版)から、各「dヌクレオチド」の化学構造式を転載します。
 Wikipedia「アデニル酸」から転載。 https://ja.wikipedia.org/wiki/%E3%82%A2%E3%83%87%E3%83%8B%E3%83%AB%E9%85%B8 |
 Wikipedia「グアニル酸」から転載。 https://ja.wikipedia.org/wiki/%E3%82%B0%E3%82%A2%E3%83%8B%E3%83%AB%E9%85%B8 |
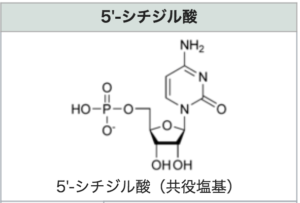 Wikipedia「シチジル酸」から転載。 https://ja.wikipedia.org/wiki/%E3%82%B7%E3%83%81%E3%82%B8%E3%83%AB%E9%85%B8 |
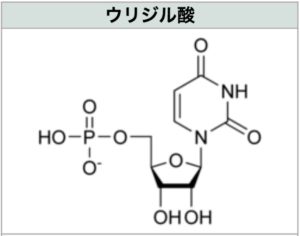 Wikipedia「ウリジル酸」から転載。 https://ja.wikipedia.org/wiki/%E3%82%A6%E3%83%AA%E3%82%B8%E3%83%AB%E9%85%B8 |
DNAの二重らせん構造のなかで、2つの「dヌクレオシド一リン酸(dヌクレオチド」が、互いに「塩基」の部分を対面させあいながら、水素結合で化学結合しています。
以下、「キリヤ化学 Q:水素結合とはなんですか?」より画像を転載します。
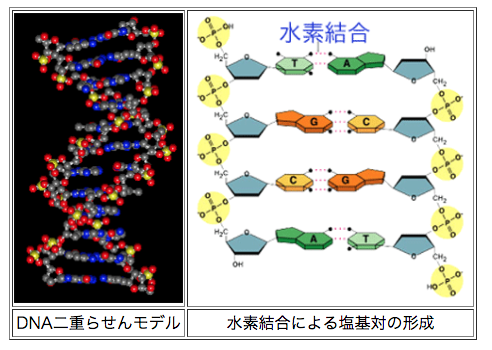
「キリヤ化学 Q:水素結合とはなんですか?」より転載。
ヒト(人間)を含む地球上のすべての生命体の生理活動は、以上みてきたような順番で、DNAの塩基配列から始まるタンパク質(立体的な4次構造)の相互作用の結果として、営まれています。
「ゲノム工学」や「合成生物学」を含む「バイオ・インフォマティックス(生命情報科学)」(は、「DNAの塩基配列から始まるタンパク質(立体的な4次構造)の相互作用」の各段階 の仕組みを、数理科学の知見とデータ解析の技法を駆使することで、解明し、人為的に編集・加工しようとする技術です。
これはまさに、情報解析・情報編集のテクノロジーであるとみることができます。
まさに、「生命情報科学」と呼ばれる所以です。
ビジネスとして立ち上がり始めたバイオインフォマティックス産業
2018年現在、バイオインフォマティックス領域は、すでに商用ビジネス産業として、立ち上がっています。
数あるデータ解析請負事業者が、遺伝子(DNA)の塩基配列を同定(「決定」と表現される)する作業である「シークエンス解析」や、(DNAの塩基配列の情報が「転写」されたmRNA(メッセージRNA)がもつ)遺伝情報のどの配列領域が活性化(「発現)し、タンパク質(アミノ酸)を生み出すのかを解析する「ポストシークエンス解析」などのデータ解析業務を有償で請け負っています。
「シークエンス解析」や「ポストシークエンス解析」など、バイオインフォマテシックスの領域で用いられている用語については、例えば以下が参考になります。
実験医学online 「オミクス」 では、これらの用語について包括的に次のように解説されています。
オミクス(omics).生体中に存在する分子全体を網羅的に研究する学問.遺伝子(gene)であればゲノミクス(genomics),転写物(transcript)はトランスクリプトミクス(transcriptomics),タンパク質(protein)はプロテオミクス(proteomics),代謝物(metabolite)はメタボロミクス(metabolomics).他にも相互作用(interaction)を網羅的に調べるインタラクトミクス(interactomics),や表現型(phenotype)を網羅的に調べるフェノミクス(phenomics)がある.
また、「トランスオミクス」 という用語については、黒田真也,中山敬一/企画『トランスオミクスで生命の地図を描け!遺伝子・タンパク質・代謝物をつなぐビッグデータ時代のサイエンス』実験医学 2014年5月号 Vol.32 No.8, 2014年04月18日発行の「企画者のことば」では、次のように概説されています。
細胞は,DNA,RNA,タンパク質,代謝物などの部品から形成される多階層にわたる大規模な分子グローバルネットワークから成り立っている.
個別の部品の一覧はゲノムプロジェクトなどにより明らかになったが,それらの部品からなる分子グローバルネットワークの全貌は同定されていない.
一方,近年急激に次世代シークエンサーや質量分析機などの技術の進展により,ゲノム,トランスクリプトーム,プロテオーム,メタボロームといったさまざまなレベルで,多階層オミクス情報を高精度かつ網羅的体系的に取得することが現実的になってきた.
これらの多階層オミクスデータを情報科学・統計数学的解析により階層縦断的に結合させて分子グローバルネットワークを同定して生体システムを解析する学問領域が 「トランスオミクス」 であり,すべての生命科学において普遍的な究極の解析方法となるポテンシャルがある.
さらに、バイオインフォマティックス領域における生体情報の解析技術についても、以下のような複数の呼称が定義されています。
- 「プロテオーム解析」:遺伝子によって生み出されるアミノ酸配列が3次元構造に折り畳まれた「タンパク質」について、それぞれのタンパク質がもつ構造と機能を解析する(別名:「プロテオミクス」)
- 「メタボロミクス」解析:着目している細胞がある期間内に産出した代謝物を解析することで、その時間内でその細胞のなかで起きた生理活動を解析する
- 「トランスオミクス」解析:ある生物の生体中に存在する分子全体を網羅的に研究する
- 「ゲノム編集技術」:本記事参照
- 「ライブ・イメージング」:生きている細胞のなかで、遺伝子の個々の塩基配列領域の発現・非発現の状況や、細胞の生理活動の様子をリアルタイムに観察・観測する技術
- 「エピジェネティック解析」:遺伝子の塩基配列それじたいは受精卵の時期から変化しないにもかかわらず、各細胞内の化学的な環境状況などの変化から受ける影響により、どの遺伝子が発現し、どの遺伝子が発現しなくなるのかの「オン・オフ」のスイッチが、細胞ごとに異なる切り替わり方で、時々刻々と切り替わる様子を解析する技術(本記事参照)
- 「インターラクトーム解析」:生きている生体の細胞のなかで、無数にある分子どうしが、互いにどのように影響を与えあっているのかを解析する技術
このように、バイオインフォマティックス領域は、記号の並び順列情報である「遺伝子の塩基配列」や、タンパク質や、それらの間の相互影響関係のネットワークの解析などを対象とする「生体情報」の「データ解析領域」としての性格を、ますます強めている、といえる状況にあります。
「DNAトポロジー」と呼ばれる研究領域もある
生体環境のなかで、生体高分子どうしがたがいに絡み合う状況を、数学の「位相幾何学」(トポロジー)の一分野である「結び目理論」の知見を用いて、絡み合いの「かたち」がもつ「情報量」(「位相不変量」と呼ばれる)を取り出して、物質の機能や挙動を分析する研究も行われています。
- 下川航也 (2016年8月) 「トポロジーと高分子」
- 河内明夫(大阪市立大学大学院理学研究科) 「結び目理論の科学への応用-プリオン分子モデルとこころのモデルを中心として」
- [スライド版] 河内明夫(大阪市立大学大学院理学研究科) 「結び目理論の科学への応用-プリオン分子モデルとこころのモデルを中心として」
この記事の冒頭でみたように、「DNA」は二重螺旋構造をしていました。

Wikipedia「二重らせん」より転載。 https://ja.wikipedia.org/wiki/%E4%BA%8C%E9%87%8D%E3%82%89%E3%81%9B%E3%82%93
このDNAが複製(コピー)されるとき、二重らせんの「結び目」構造をもつDNAは、「結び目」が局所的にほどかれていきます。
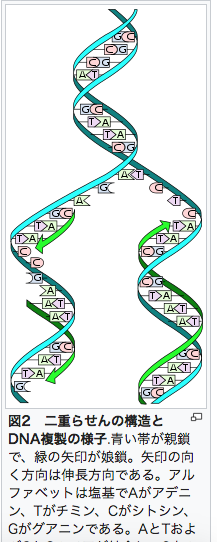 Wikipedia「DNA複製」より転載。 https://ja.wikipedia.org/wiki/DNA%E8%A4%87%E8%A3%BD |
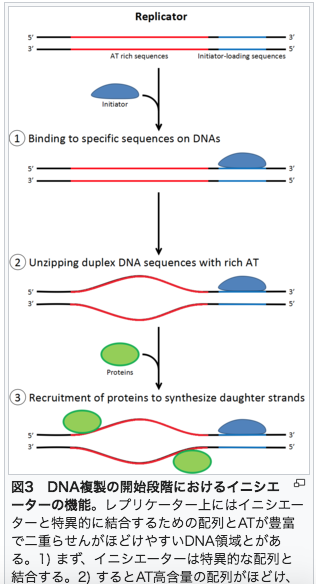 Wikipedia「DNA複製」より転載。 https://ja.wikipedia.org/wiki/DNA%E8%A4%87%E8%A3%BD |
その際、DNAの「二重らせん」構造を、絡まって結び目ができないようにしながら、どのようにしてほどいていくのかのメカニズムを、数学の「結び目理論」を用いることで解明することができたという、研究成果も出ているようです。
- とね日記 (2015年07月12日 19時45分36秒)「多次元空間へのお誘い(14):DNAの複製について」
- De Witt Sumners, Lifting the Curtain: Using Topology to Probe the Hidden Action of Enzymes, NOTICES OF THE AMS(American Mathematical Society), VOLUME 42, NUMBER 5
- Sergei M Mirkin, DNA Topology: Fundamentals, ENCYCLOPEDIA OF LIFE SCIENCES / & 2001 Nature Publishing Group / www.els.net
- 下川 航也 「結び目と数学とDNA」
また、Gigazine(2018年04月25日 15時00分)「サイエンス 生きたヒト細胞内で初めてDNAの結び目構造「i-motif」が確認される」にも、注目が集まっています。
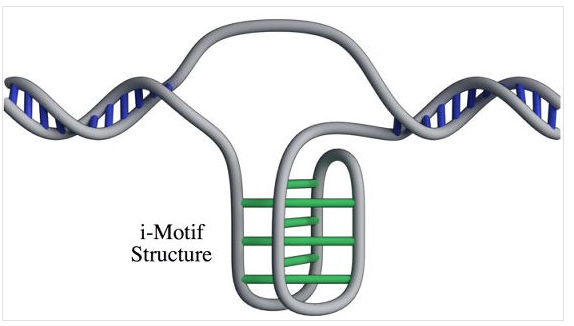
Gigazine(2018年04月25日 15時00分)「サイエンス 生きたヒト細胞内で初めてDNAの結び目構造「i-motif」が確認される」より転載。
DNAには二重らせん構造以外にも「i-motif」や「G-quadruplexes(グアニン四重鎖)」などの特殊な構造を持つものが知られています。一般的なDNAは、塩基のアデニン(A)とチミン(T)が、シトシン(C)とグアニン(G)が結合して二重らせんを形成するのに対して、DNAの4本鎖の「結び目」に例えられるi-motifではC同士が結合して特殊な構造を作り出すとのこと。
今回、特殊な構造である「i-motif」を持つDNAを、Garvan Institute of Medical Researchのダニエル・キリスト博士たちの研究グループが、生きたヒト細胞内で観察することに成功しました。これまでは研究室で人工的に作り出した条件の下でのみi-motif構造を観察することができていましたが、自然なヒト細胞内で観察されたのはこれが初めてだそうです。
研究者たちはヒト細胞内のi-motifを特定するために、特定の物質と結像する性質を持つ「Y型」の分子を使って小さなプローブを作りました。このプローブはi-motifのみに結合し、他の形態のDNAには結合しない特殊なものだとのこと。蛍光色素を添加したプローブを使って異なる3種類のヒト細胞を調べると、緑色の斑点が現れては消えるのが確認され、i-motifの形成と再形成が確認されました。
「結び目理論」の知見が、バイオ・インフォマティックスを含む物質科学や、宇宙物理学などのその他の物理学において活用されている状況については、以下のスライドで概観されています。
「結び目理論」については、以下が分かりやすいです。
タンパク質立体構造予測コンテスト: CASP
遺伝子の塩基配列の記号の並び順が指定するアミノ酸から、リボソームにおいて、どのような立体構造のタンパク質が生成されるのかを予測する課題は、「タンパク質構造予測問題」と呼ばれています。
タンパク質の形状(構造)が、データ解析によって予測すべき主題となるのは、あるタンパク質がもつ立体構造の形状が、そのタンパク質が生体組織のなかでどのような機能を担うのかを決める因子となると理解されているからです。以下、Wikipedia(日本語版)「タンパク質」の一節を引用します。
立体構造の決定 上記のようなタンパク質の高次構造は、X線結晶構造解析、NMR(核磁気共鳴)、電子顕微鏡などによって測定されている。
また、タンパク質構造予測による理論的推定なども行われている。
タンパク質の立体構造と機能は密接な関係を持つことから、それぞれのタンパク質の立体構造の解明は、その機能を解明するために重要である。
いずれ、ほしい機能にあわせてタンパク質の立体構造を設計し、合成できるようになるだろうと考えられている。
これまでの研究により構造が解明されたタンパク質については、蛋白質構造データバンク[12]によりデータの管理が行われており、研究者のみならず一般の人でもそのデータを自由に利用、閲覧できる。
- 河野圭祐ほか「タンパク質二次構造予測を行う深層学習モデルの Saliencyによる可視化」, The 31st Annual Conference of the Japanese Society for Artificial Intelligence, 2017
- 渡邉 和之・三枝 亮・橋本 周司「ニューラルネットワークによるタンパク質立体構造予測の試み」, 情報処理学会第69回全国大会
- Seamless (2018/12/04)「Google DeepMind、アミノ酸配列から機械学習を用いてタンパク質の立体構造を推定する「AlphaFold」発表。難病の原因解明や治療法に役立てる」
- Wired(2011.09.21 WED 11:48) 「難問のタンパク質構造をゲーマーが解析」
- 朴 聖俊ほか「タンパク質立体構造予測の現状と未来」, 人工知能学会論文誌 20 巻 4 号 0512(2005 年)
タンパク質の立体構造予測問題とは,与えられたアミノ酸配列をもつタンパク質が生体内でとる三次元立体構 造を理論的に予測する,という至ってシンプルな設定の問題である.
構造予測研究は 40 年にもわたる長い歴史 をもち,多くの挑戦と失敗とを繰り返しながら,徐々に 理論と実践の両面から成熟してきた. 現在の構造予測研究の最先端では,物理化学とバイオインフォマティクスを融合させており,これまでに蓄積 してきた物理化学の側面と情報学の側面を巧みに活用している.
そして,場合によっては X 線構造と同レベルの予測が可能になり実用化されるとともに,タンパク質の 構築原理を理解するための基礎研究の一つとなっている.
この分野の現状は,1994 年以降 2 年に一度行われている構造予測技術評価会議 CASP(Critical Assessment of Techniques for Protein Structure Prediction)の結果から非常によく概観できる.
本解説は,タンパク質立体構造予測における最新の研 究成果と動向について,2004 年に行われた CASP6 を一 望することによって理論と実践の両面から解説し,構造ゲノム科学における応用と期待について述べる.
この「タンパク質構造予測問題」に対して、DNAの塩基配列の記号の並び順を学習データとしたデータ解析の予測モデルを用いて予測する精度を競い合う国際競技会としては、CASP(「構造予測技術評価会議 」)という競技会が開催されています。

(参考)
CASP: Google DeepMindの出展モデル
この技術競技会には、Google DeepMindもAIを駆使した予測モデルを出展しています。
DeepMindが出展したモデルについては、AInowから以下の記事が出ています。これは、DeepMindの技術ブログを和訳したもののようです。
本記事の構成
この記事では、まず最初に、今日の遺伝子編集工学・合成生物学が、文字としての遺伝子を対象とする文章解読・編集技術として、どこまで発展しているのかを概観します。
次に、記号の並び順序が担う意味を解読・編集する自然言語処理技術をはじめとする人工知能技術が、そのような「文字としての遺伝子を対象とする文章解読・編集技術」としての遺伝子編集工学・合成生物学の発展をどのように加速化させているのかを確認していきます。
1. 人類は、塩基を文字にもつ「ゲノム言語」をどこまで、操れるようになったのか?
ゲノム工学の現状:遺伝子編集技術と遺伝子合成技術
今回の記事では、カーツワイル氏による『ポスト・ヒューマン誕生』の刊行後に、生きている生物がもつ遺伝子(塩基配列)の狙った位置の文字列(塩基配列)を、人類が、1文字(1塩基)から任意の長さの文字数(塩基数)だけ、正確に編集・改変することを可能にするゲノム編集技術が、大きく発展した事実を見ていきます。
後に見るように、2012年6月に発表されたある論文によって、CRISPR-Ca9という遺伝子編集ツールが提案されたことが、この領域の技術を飛躍的に前進させる結果となりました。
(以下、Wikipedia日本語版「ゲノム編集」所収の遺伝子のイメージ図を転載)
Wikipedia日本語版「ゲノム編集」所収の遺伝子のイメージ図を転載。
続いて登場したのは、人類が、自然界にすでに存在する塩基配列に手を加えて編集・加工を施すのではなく、人類がなにもない「まっさらな白紙の状態」から、「塩基」という「文字」を1文字ずつ書き下していくことで、これまで自然界に存在しなかった、まったくあらたな「文章」(塩基配列=生命体の設計図)を創作することを可能にする技術です。
「塩基」どうしを人為的に化学結合させることで、まったく新しい塩基配列を生み出すこの技術は、「合成生物学」(Synthetic biology)と呼ばれています。
この「合成生物学」の技術を用いて、人類は、わずか500個未満の塩基配列で生成される「生命活動を営むことができる」人工生命体を、遺伝子レベルで初めて人工的に作り出すことに、すでに成功しています。
クレイグ・ベンダーという合成生物学者が生み出したこの「人工生命体」は、生命活動を行うことができる能力をもった生物は、最低・何文字の塩基配列でつくりだすことができるか?という問いに答える探求のなかで作り出されたものです。
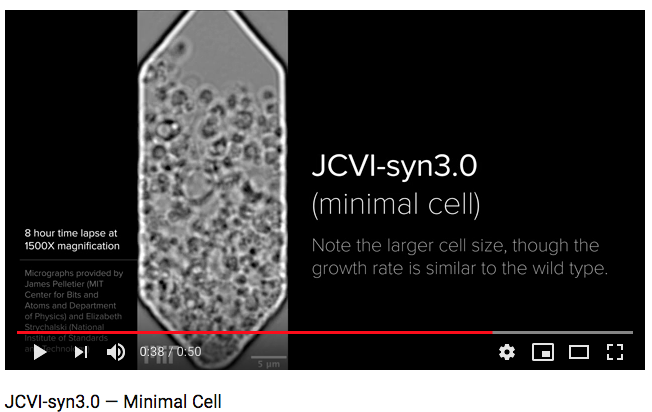
J. Craig Venter Instituteが公開した人類史上初めてとなる遺伝子の化学合成によって人工的につくられた生命体 JCVI-syn3.0の姿を捉えたYouTube解説動画
ベンダー氏が、「まっさらな白紙の状態」から生み出したこの「人工生命体」は。これまで人類が知り得たすべての「生きることのできる」生物のなかで、もっとも短い文字数の塩基配列をもつ生命体であるため、「ミニマム・セル」(最小サイズの細胞)とも呼ばれています。
これは、「合成生物学」の知見と技術を用いることで、人間が人工的に新種の生命体を作り出すことができるという事実を、世界で始めて実証した新時代を切り開いた出来事として、広く知られています。
カーツワイルがGNR論で展望していた、遺伝学によるヒト人体の強化を実現させるための国際プロジェクトも、すでにアメリカで動き始めています。
アメリカ国防総省・国防高等研究計画局(DARPA)が資金的に後押しをしている「Genome Project–Write (GP-write)」計画がそれです。
このプロジェクトは、人間の遺伝子である「ヒトゲノム」の塩基配列を人為的に「書き換える」(Re-write)ことで、近い将来、疫病への感染や過酷な自然環境に耐性をもつように、人体を改修する(強化する)ことを可能にするための要素技術を獲得することを目指すものとされています。
Genome Project–Writeの事務局公式ウェブページのトップページ
遺伝学(ゲノム工学)は、当初、自然界に存在する既存の生命体がもつ遺伝子を対象として、数文字から数百文字の塩基配列を部分的に削除したり、挿入したり、交換によって書き換えることを目的にしていました。遺伝性の疾患の原因となる特定の遺伝子の特定の塩基部分(変異部分など)に焦点をあてて、疾患を引き起こしている1文字から数百文字の塩基(配列)を、ピンポイントで修復することで、遺伝病を根治させたり、害虫に強い食用の作物を生み出すことが、その実用的な活用法として設定されていました。
その後、この遺伝学(ゲノム工学)から、あらがな学問分野として発展してきた「合成生物学」は、 自然界にすでに存在する遺伝子に、人為的に部分的な改修の手を加えるという発想ではなく、まったく白紙の状態から、人間が塩基を化学的に結合させて、つないでいくことで、自然界にこれまで存在しなかったあらたな塩基配列をうみだすことを目標に掲げています。これはいわば、「塩基」という「文字」で、最初の1文字目から新たな文章を創作していくような営みです。
さらに、日本の理化学研究所は「塩基」という「文字」そのものを、人為的に新しくつくりだすことに成功しています。(人間による、新たな「文字」の出現)
これまで地球上で発見されたすべての生物の細胞は、4種類の塩基(A:アデニン、T:チミン、G:グアニン、C:シトシン)の4種類の塩基から構成される20種類のアミノ酸によって構成されていました。これまで発見された生物の細胞を構成するタンパク質はすべて、これら自然界に存在する20種類のアミノ酸が、様々な構造で化学結合をすることで織りなされていました。
理化学研究所が人工的に作り上げた2つの塩基が、これまえで自然界に存在していた4つの塩基にあらたに加わることで、地球上には、合計6つの塩基が存在するようになりました。これら6つの塩基を組み合わせることで、これまで地球上に存在しなかった新種のアミノ酸を生み出すことが理論的に可能であり、これら未知のアミノ酸を組み合わせることで、さらに新種の人工的なタンパク質を作り出すことに道を切り開くことが期待されています。
これら人工の塩基、人工のアミノ酸、人工のタンパク質が、生きた生物の細胞環境のなかで、どのような条件のもと、どのような挙動を示すのかは、今後の研究の課題となるものと思われます。そこで得られた知見をもとに、6つの塩基が織りなす新種のアミノ酸・新種のタンパク質を用いて、病気に効く薬を生み出したり(創薬)、この記事の本文でみていくように、産業的に有用な物質やエネルギーを精製する人工細胞(スマート・セル。Smart cell)を人工的に構築していくことが期待されています。
これはまさに、カーツワイル氏が『ポスト・ヒューマン誕生』で描いていたゲノム工学(G: 遺伝学)の進歩による人体と、人間が暮らしを営む上で、資源として活用することのできる「生物資源」の自由な作り変え・再編集を可能にするシナリオです。
人工細胞に自己複製・自己修復が可能な産業資材を精製させる「合成生物学」
「合成生物学」(Synthetic Biology)は、これまで人類が獲得してきたものづくりの技術である機械工学や電子工学、有機化学工学では生み出せなかった以下の挙動(機能)をもつ材料資源(資材)を生み出すことが期待されています。
- 自己複製が可能な資源(資材)
- 自己修復が可能な資源(資材)
- 環境の変化に自律的に適応することが可能な資源(資材)
記事の本文で取り上げるように、我が国の経済産業省はすでに、人工細胞を「ものづくり」の担い手(「工場」)として利活用する「スマート・セル・インダストリー(Smart Cell Industry)」という概念のもと、どのようなあらたな産業を興すことが可能なのかを、我が国の産業戦略を立案するための基礎資料としてとりまとめています(ウェブサイトで公開されているスライド資料です)。
人間の肉体・知性の「強化」を目指す「合成生物学」
「合成生物学」はまた、カーツワイル氏がそのGNR論のなかで将来展望として描いたように、生物としてのヒトの肉体(と知性)を、ゲノム工学の力を用いて「強化」する方向性でも、すでにその道を歩み始めています。
米国防総省の高等国防研究計画局(DARPA)が資金提供を行うヒトゲノムの書き換え(re-write)プロジェクトがそれです。そこでは、感染症ウイルスに対して耐性をもつように(現行の)ヒトゲノムを部分的に改修(改善)させたり、過酷な生活環境で長期間、安定的に活動できるように(現行の)ヒトゲノムを部分的に改修(強化)させることが、将来的な目標として掲げられています。
遺伝子工学:ヒトゲノム配列の98パーセント以上が担う機能は、まだ未解明である
塩基配列という「記号体系」に秘められた文法構造の解読作業については、(m-RNAにおける)3文字の塩基配列(「コドン」と呼ばれる)が特定のアミノ酸の組成を指定する暗号表(「コドン表」)はすでに解読されていますが、ヒトゲノム(人間の全塩基配列)のうち、アミノ酸の組成を指示している部分(「ヒストン」と呼ばれている領域)は2%弱しかなく、ヒトゲノムの塩基配列のうちの98%以上は、個々の「コドン」の発現度合いや発現条件を指定していることが徐々にわかりはじめているものの、それが担う「意味」(文法構造)は、いまだ未解明なのです。
これらを「解読」する上で、近年発展著しい数理統計学や(ディープ・ラーニングを含む)機械学習の「情報解読技術」は、どのように活用されているのでしょうか?
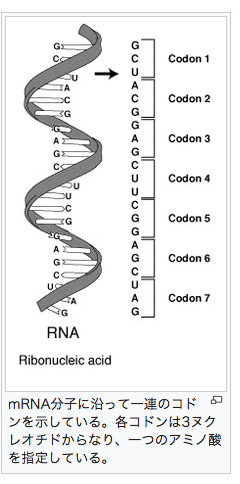
Wikipedia 「コドン」より
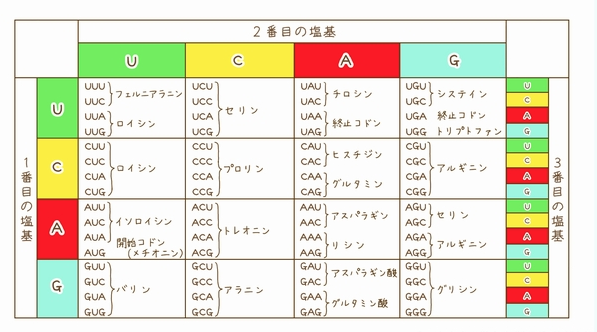
(以下、日本生物工学会「『ひらく、ひらく「バイオの世界」』イラスト・写真館 ―コドン表の読み方(Q8)―」より、コドン表の解説図表を転載)
遺伝子工学:ゲノムの後天的な修飾(エピジェネティクス)の仕組みは、まだ未解明な部分が多い
また、ヒトゲノムは、人間の身体の機能のすべてを、あらかじめ決定論的に指定する能力はもっておらず、(同じ塩基配列をもつ)一卵性双生児であっても、双生児の兄弟の兄と弟(あるいは、姉妹における姉と妹)は、それぞれ、誕生後に経験する物事が違います。
この誕生後に体験する行動環境と「からだ」とのフィードバックの履歴(いろいろな体験のなかで感じる様々な「ストレス」の履歴を含む)によって、兄と弟の体細胞の中では、スイッチが「オン」になる「塩基配列」の「部分」と、スイッチが「オフ」になる「塩基配列」の「部分」が異なるようになります。
こうして、完全に同じ塩基配列を持った一卵性双生児の兄弟・姉妹であっても、時間を経るごとに、塩基配列上の「オン」・「オフ」の並び順の違いが大きくなっていくことで、(塩基配列は同じままでありながらも—ただし、特定の塩基の突然変異などは除く)一卵性双生児の兄(姉)と弟(妹)の顔や身体の外観も性格も、成長に従って次第に違う肉体・人格になっていくことが知られています。
この現象が発生するメカニズムは、遺伝学において、「エピジェネティクス」(Epigenetics)と呼ばれていますが、ヒトゲノムのうち、塩基配列の「ある部分」のスイッチが「オン」になったり、「オフ」になったりする条件に関しては、行動環境のどのような要素が原因となっているのかや、摂取する栄養条件のどれが原因となっているのか、まだまだ未解明な部分が多いのです。
3つのレベルで、人類は「ゲノム言語」を操り始めている
カーツワイル氏の『ポスト・ヒューマン誕生』が出版された後、人類は、次に示す3つのレベルで、「ゲノム言語」を操り始めているということができます。
- 自然界にある既存の塩基配列を修正・改変する「遺伝子編集技術」:CRISPR-cas9技術とその後継技術や、「遺伝子ドライブ技術」
- 既存の塩基配列の修正・改変ではなく、人間がゼロから「塩基」の文字で新しい文章を書いていく「合成生物学」
- 人間が、ゲノム言語の文字である「塩基」そのものを新しく作り出す技術(人間が、ゲノム言語に新たな「文字」を追加する技術)
以下、上記の3つについて、ひとつひとつ見ていきます。
(レベル1) CRISPR-cas9技術の登場:塩基「文字」単位の遺伝子改変を、数万円・数日間の費用で正確に行うことが可能になった
遺伝子改変技術:2000年代以前の状況
自然界にすでに存在する遺伝子(塩基配列)を対象として、標的とする一部の塩基部分配列を削除したり、書き換えたりする技術は、分子生物学において、長年とりくまれてきたテーマでした。
1990年代までの遺伝子改変技術は、生きた生物の細胞の塩基配列に、目的とする塩基配列を挿入する場合に、その細胞がもつ細胞膜を通り抜けて細胞内の奥深くの細胞核まで入り込むことのできるウイルスを利用する方法が広く用いられていました。
目的とする塩基配列(DNA断片)を入れたウイルス(「運び屋」(ベクター)と呼ばれる)に、対象とする生物の狙った細胞の細胞核まで、DNA断片を運んでもらおうというアプローチなのですが、ウイルスが対象とする生物の細胞核にうまく入り込むことに成功しても、対象生物がもつ塩基配列のなかの標的にする部分に、正確に(ウイルスが運び込んだ)DNA断片を挟み込むことが困難でした。
つまり、遺伝子改変の正確性が、まだまだ低かったのです。その上、この手法を実行するには、分子生物学やウイルス学に関して深い見識と経験をもった熟練した研究者の手を借りる必要があり、さらに、ウイルスに運び込んでもらいたいDNA断片を人工的に作り出すにも、高額にのぼる費用と作成期間が必要でした。
遺伝子改変技術:2000〜2012年半ばまでの状況
須田 桃子氏著『合成生物学の衝撃』(文藝春秋社刊行)の55ページ目によると、「2000年代後半になり、ゲノムを狙い通りに改変できる『ゲノム編集』という技術が開発された。ゲノムの目的の塩基配列を探し出すガイド役のタンパク質と、その部位を切断するハサミ役の酵素の酵素を組み合わせて細胞に送り込む技術で、『編集』という言葉の通り、遺伝子改変の精度が格段に上がった。ただし、ガイド役のタンパク質を作成するのに手間とコストがかかるのが何店で、使われる研究は限られていた」という状況でした。
遺伝子改変技術:2012年6月以降の状況(改変の精度もコストも飛躍的に改善された)
しかし、こうした状況は、2012年6月に、米国・カリフォルニア大学バークレー校のジェニファー・ダウドナ教授とスウェーデン・ウメオ大学のエマニュエル・シャルパンティエ教授が発表した論文で提唱されたCRISPER-Cas9という手法によって、大きく乗り越えられることになりました。
同書の同じ55ページを続けて引用すると、「タンパク質の変わりに作成の簡単なRNAをガイド役にしたこの技術は、効率と精度に優れ、おまけに低コストと三拍子そろっていたことから、爆発的に世界中の研究者に普及した」ということです。
CRISPER-Cas9:その由来は、細菌がウイルスに対してもつ免疫機構である
なお、このCRISPER-Cas9という名前は、CRISPERという文字列と、Cas9という文字列の2つで構成されています。
まず、CRISPERという文字列は、もともと細菌がもっていた塩基配列のある特徴的な部分を指す用語です。
その特徴とは、前から読んでも後ろから読んでも同じ文字列に読めるという性質です。このような文字列を、(ゲノム工学や分子生物学に限らず、ひろく一般に)「回文」と呼ばれています。
先程、名前を挙げた米国・カリフォルニア大学バークレー校のジェニファー・ダウドナ教授は、細菌が(自分の体のなかに侵入する)ウイルスをどのように検知して、撃退しているのかという、細菌が持つ免疫のメカニズムを研究していた研究者でした。
ダウドナ教授は、細菌のDNA(塩基配列)のなかで、「回文」の特徴をもつ塩基配列の領域に挟み込まれた領域に、その細菌の先祖たちが、過去に感染した経験のあるウイルスの塩基配列の断片が埋め込まれていることを突き止めたのです。
次に、Cas9ですが、Cas9は、細菌自身のDNA(塩基配列)のなかでの「回文」(CRISPER)が出現する領域の付近に存在する塩基配列を指す言葉です。このCas9と名付けられ部分の塩基配列が生み出すのは、ある「酵素」を生み出します。その「酵素」は、ある「ウイルス」DNAをズタズタに切り裂く「ハサミ役」の役割を果たすものです。
以上、要約すると、Cas9は、この「回文」領域の付近に出現する(「ハサミ役」の)酵素の生成を指示する塩基配列部分の名前なのです。
細菌がウイルスに対してもつ免疫機構の仕組み
細菌は、あらたに自分の体内になんらかの異物が侵入したことに気がついた場合に、その「異物」が持つDNAが、自分(細菌)自身のDNAのなかの回文(領域)と回分(領域)で囲まれた領域に埋め込まれている塩基配列の断片と一致するかどうか照合し、一致する場合は、その「異物」が、過去に自分のご先祖様に感染したのと同じ種類の「ウイルス」であることを認知して、「ウイルス」であるその「異物」を撃退しようと動きだすことを明らかにしました。
この前から読んでも後ろから読んでも同じ文字列になる「回文」構造をした塩基配列のことを、「リピート配列」(または「反復配列」)と呼びます。
また、2つのスペーサー配列によって挟み込まれた、ウイルスの塩基配列の一部分が埋め込まれた領域の塩基配列は、「スペーサー配列」と名付けられています。
具体的な手順はこうです。
まず、新しいウイルスが自分(菌)の細胞のなかに入り込んだときに、ウイルスの二重らせんの形をしたDNAを、CAS9とよばれる酵素を用いて、「ファスナーをほどくように」こじあけて、ウイルスがもつDNAの塩基配列を読み取れるようにします。
次に、”自分(細菌)自身のDNAのなかの回文(領域)と回分(領域)で囲まれた領域に埋め込まれている塩基配列の断片”を、DNAからRNAに「転写」することで、単体のRNAを作り出します。
そして、先程こじあけたウイルスの塩基配列のなかに、作り出したRNAと文字列が完全一致する部分があるかどうかを、照合していきます(UNIXコンピュータの文字列検索であるgrep処理と同じです)。
そして、文字列が完全に一致する部分が見つかった場合は、そのウイルスは、過去にご先祖様の身体(細胞)を襲ったことがあるウイルスであると認識し、(先程、ウイルスの塩基配列を読み取れるように、ウイルスのDNAの2重らせんをほどくのに用いた)CAS9とよばれる酵素をつかって、そのウイルスの塩基配列をバラバラに切り刻むことで、ウイルスを殺傷・無力化するのです。
上記のプロセスについては、以下の2つの解説が参考になります。
- 「原核生物の新規な獲得免疫機構CRISPR/Casシステム リピートとスペーサーの規則的反復構造が細菌の進化をコントロールする」, 化学と生物, 2013年
- 京都大学大学院医学研究科・微生物感染症学分野「CRISPRとは」
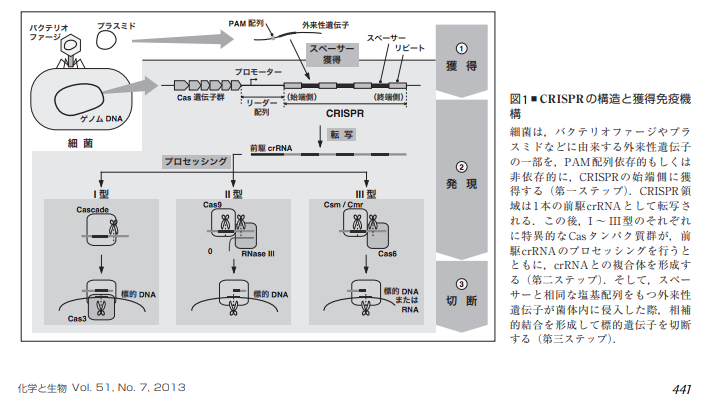
化学と生物 Vol. 51, No. 7, 2013 所収の解説資料(1)より図版部分を抜粋して転載。
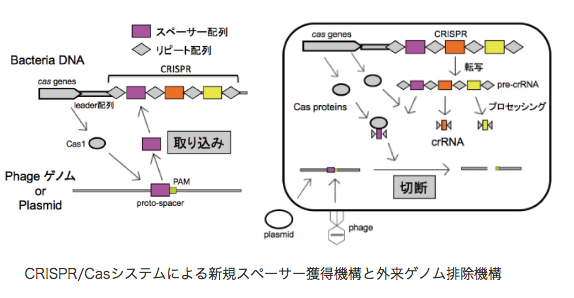
京都大学大学院医学研究科の解説資料(2)より図表部分を抜粋して転載
ダウドナ教授が率いる研究所 Doudna Lab
ダウドナ教授は現在、Doudna Lab研究所を率いており、以下に述べるようなCRISPR-cas9のさらなる改良化に向けた研究活動を牽引されています。
Doudna Lab.のwebサイトのトップページ
細菌が持つ免疫機構を取り出して、人間による遺伝子編集ツールとして活用するのが、CRISPER-Cas9キット
CRISPR-Cas9は、細菌が持つDNA(塩基配列)の2つの「回文」ではさみこまれた領域に書き込まれていた「塩基配列」を、人間が、「目的とする塩基配列の文字列」を挿入したり、上書き書き換えさせたいと考えている、標的生物がもつ「標的とする塩基配列」に書き換えたものです。
先程の須田氏の著書の55ページを引用すると、「タンパク質の変わりに作成の簡単なRNAをガイド役に」することで、人間が標的に定めた部位(塩基配列の特定の塩基領域)に、正確に、「目的とする塩基配列の文字列」を運んで、その位置にもともともあった塩基配列の前後に挿入したり、もともとあった塩基配列と交換して、塩基配列を上書き更新させることが可能となったのです。
このCRISPR-Cas9の登場で、遺伝子改変の実行は、熟練の研究者や技術者がいなくても、狙い定めた標的部分を正確に遺伝子編集することができるようになり、しかも短期間に、これまでと比べて圧倒的に安価な費用で行う事が可能になりました。
sgRNAライブラリの登場
このように、CRISPR遺伝子とcas9遺伝子は、もともと、菌類がウイルスを撃退するためにもっていたものですが、この「CRISPR」部分と「cas9」部分を、人間が、遺伝子を削除したり書き換えるために利用しやすいように改変した遺伝子編集用のツールが、sgRNAライブラリです。
Google検索エンジンで、「sgRNA」という文字列で検索をかけると、例えば以下のウェブページがヒットします。
CRISPR-cas9を使って、遺伝子書き換えが実行された生物のリスト
(作成中)
CRISPR-cas9の後継手法
CRISPR-cas9には、まだまだ改良すべき点があるという報告が寄せられています。
- Wired誌オンライン版記事(2017.07.05 WED 09:30付け) 「ゲノム編集技術『CRISPR』は“終わった”のか──たったひとつの論文から始まった風評と、検証なき「情報共有」の危うさ」
以下のWired誌の日本語版の記事が参考になります。
(レベル2) 既存の塩基配列を編集することで、必要なゲノムを獲得するのではなく、人間が白紙の状態から、「塩基」の文字でゲノム配列を書いていく「合成生物学」(Synthetic biology)
CRISPER-Cas9は、(自然界が生み出した)既存の塩基配列を編集することで、必要なゲノムを獲得する技術でいた。
これに対して、(自然界が生み出した)既存の塩基配列を編集することで、自然界にこれまでになかった塩基配列を人間が生み出すというアプローチを取るのではなく、人間が白紙の状態から、「塩基」の文字でゲノム配列を書いていく方向性(アプローチ)を目指すのが、「合成生物学」(Synthetic biology)です。
「生命体」を設計する上で、必要最小限の塩基文字数を探り当てる試み:「ミニマム・セル」プロジェクト
クレイグ・ベンダー氏が率いるJ. Craig Venter Institute(クレイグ・ヴェンター研究所)は、自然界に存在する人類にとって既知のあらゆる生命体のなかで、その塩基配列(のゲノム)の数が最も少ない生物として認知されていたマイコプラズマ(mycoplasma)よりも、さらに少ない、わずか473文字の塩基配列を持った人工生命体を作ることに成功しました。
自然界にすでに存在するマイコプラズマ菌の写真画像。 英語版Wikipedia Mycoplasma より転載。 (Source) https://en.wikipedia.org/wiki/Mycoplasma#/media/File:M._haemofelis_IP2011.jpg
JCVI-syn3.0と名づけられたこの「人工生命体」は、人間が、塩基配列を一から化学合成(人工合成)することで生み出すことに成功した、生命活動を営む能力を宿した生命体として、知られています。
(J. Craig Venter Instituteが公開したJCVI-syn3.0の姿を捉えたYouTube解説動画)
- J. Craig Venter Instituteが2016/03/24 に公開 “JCVI-syn3.0 — Minimal Cell”
ベンダー氏らによるこの試みは、Minimum Cell Projectと命名されたものです。
クレイグ・ベンダー研究所のプレス・リリースはこちらになります。

以下はプレス・リリースのPDF版になります。

また、論文としては、以下があります。
- Hutchison CA et.al. , Design and synthesis of a minimal bacterial genome., Science (New York, N.Y.). 2016-03-25; 351.6280: aad6253.
- Roy D. Sleator, JCVI-syn3.0 – A synthetic genome stripped bare!, Bioengineered. 2016 Mar-Apr; 7(2): 53–56.
ベンダー氏は、自身が率いたミニマム・セル・プロジェクトについて、TED Talkにも登壇してプレゼンテーションを行っています。
カーツワイル財団も、2016年3月28日付の記事“Craig Venter’s team designs, builds first minimal synthetic bacterial cell”で取り上げています。
また、Nature誌も、以下の記事を掲載しています。
クレイグ・ベンダーは、世界で初めて、白紙の状態から人間が塩基どうしを結合させて、生きる能力をもつ「生命体」を生み出した
このように、「わずか473文字の塩基配列で書かれた」『「生きることができる」人工生命体』が生み出されたことで、人間がまったくの白紙の状態から、塩基(文字)を1文字ずつ、人為的に書き連ねる(塩基どうしを化学的に結合させあう)ことで、実際に「生命力」を宿した生命体を生み出すことができるということが、実証的に証明されたことになります。
ミニマム・セル・プロジェクトの関連ウェブページ及び論文
- (レビュー論考)Anthony C Forster & George M Church, Review Towards synthesis of a minimal cell, Molecular Systems Biology (2006) 2, 45
- Jewett MC & Forster AC., Update on designing and building minimal cells., Curr Opin Biotechnol. 2010 Oct;21(5):697-703.
ヒトゲノムの遺伝子書き換えを目指す国際プロジェクト: Genome Project–Write (GP-write)計画
ヒトゲノムの遺伝子書き換えを目指す国際プロジェクトとして、「Genome Project–Write (GP-write)計画」が進行しているようです。これは将来的には、病原菌やウイルスに対して耐性をもつように、現行の人間(ヒト)の生体を強化するといった長期展望を見据えていることなどがささやかれています。
(以下、Genome Project–Writeの事務局公式ウェブページのトップページを転載)
このプロジェクトについては、須田 桃子(著)『合成生物学の衝撃』(文藝春秋社から、2018年4月13日に刊行)で、CRISPR-cas9の発見とモデル動物による概念実証を世界に先駆けて行った研究者とのインタビューを交えて、深く論じられています。

ここでは、同プロジェクトについての情報をまとめたウェブページであるSCIENCE CRIAP_BIO (2018年5月2日付け)「ゲノムを書くプロジェクト ‘GP-write’は,「ヒト遺伝暗号の書き換え」から始まる」から、以下の関連文書と記事へのリンクを転載します。
- Scientists downsize bold plan to make human genome from scratch. Dolgin E. Nature2018 May 1.
- Genome ‘writers’ set their first goal: recoding human cells to resist viruses. Begley S. STATS News2018 May 1.
- GP-write Announces ‘Ultra-safe Cells’ as Featured Community Project. GP-write BLOG
Smart cell industryという概念:生物細胞に、産業上、有用な物質を「生産させる」という発想
合成生物学の技術を用いることで、人間によって有用性の高い生物細胞を、人為的・人工的に生み出そうとする試みは、世界中で、すでに多くの数の計画プロジェクトが走り始めています。
個々の計画プロジェクトの目的設定は異なります。個々のプロジェクトの目的ごとに、「有用性の高い生物細胞」という場合の「有用性」の定義は異なるのです。
「有用性」の例としては、例えば、以下があります。
- 製薬産業の創薬事業における特定疾患に効能のある医薬品の開発
- 農業事業における病原菌やウイルスや悪天候や水不足に強い農産物の開発
- エネルギー産業におけるバイオ燃料を効率的に生み出す生物細胞の開発
- 環境保護団体における環境を浄化・回復させる環境効果を発揮する生物細胞の開発
このように、産業上・経済上の富(売上・利益)を生み出すことを目的とした合成生物学のビジネス利活用がの(産業)競争がすでに始まっています。
あらたな市場における市場シェアの獲得をめぐる熾烈な産業競争の火蓋がすでに切られており、潜在成長力が高いと評価されたバイオ・ベンチャー企業には、世界中の機関投資家や個人投資家やベンチャーキャピタルから、数十億円〜数千億円規模のリスク・マネーが、投下資本として投じられています。
こうした中、産業上、有益な資源を産出する「生物細胞」を生み出す営みは、「スマート・セル・インダストリー」と呼ばれ始めています。以下は、我が国の経済産業省の資料です。
経済産業省の上記のスライドから、スライドをいくつか抜粋して転載してみます。
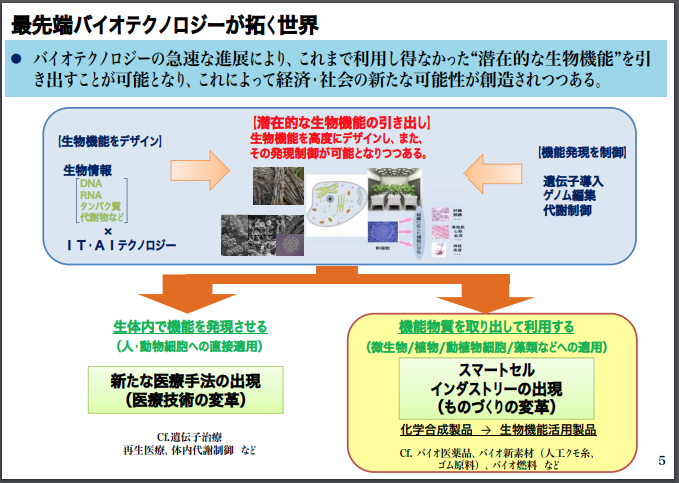
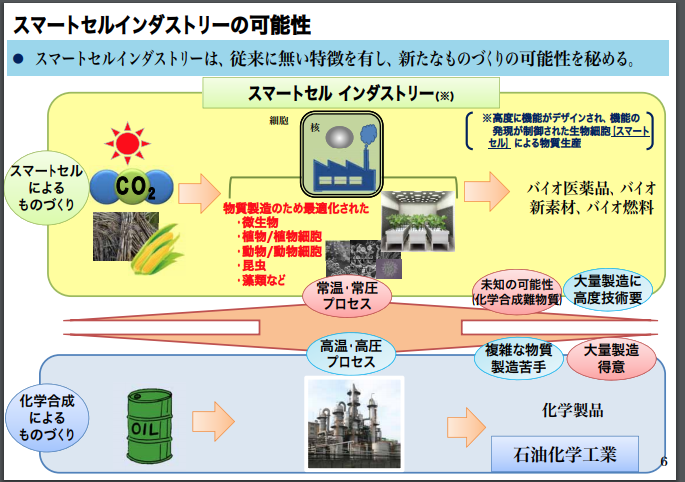
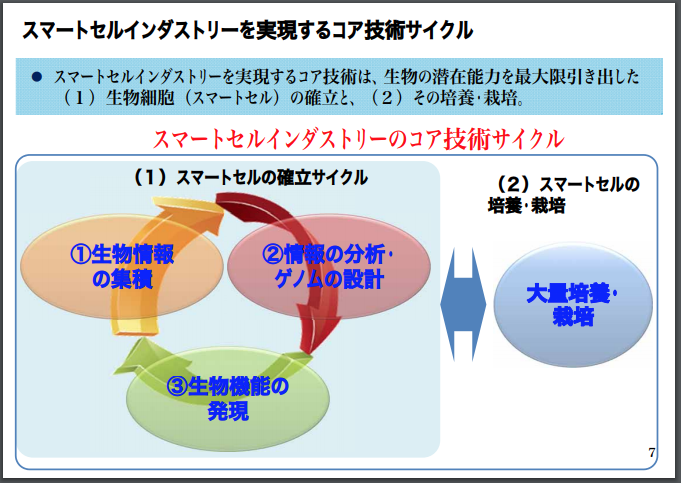
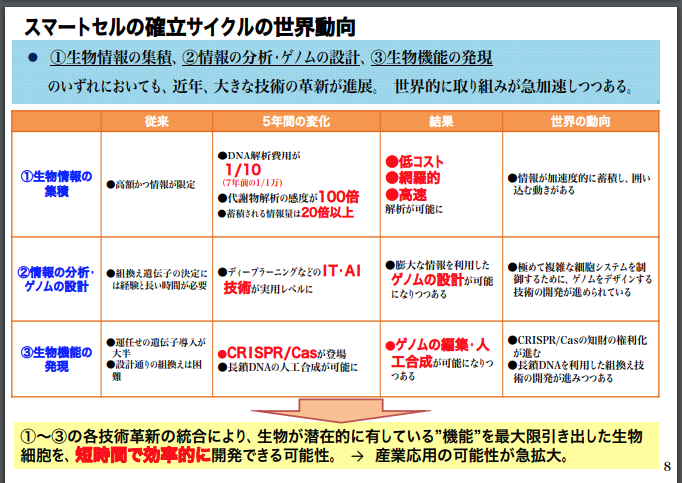
最後の4枚目のスライドでは、合成生物学の知見を用いて、目的とする産業上、「有益な資源」を「生産する」能力をもつような「生物細胞」をつくるためには、どのようなゲノム設計情報(どのような塩基配列)が有効であるのかを解探索する上で、A.I.人工知能の計算技術を活用することが有望であると、書き込まれています。
このあたりは、ゲノム工学・合成生物学とA.I.(人工知能)技術の融合技術・複合技術について論じていく際に、再び立ち戻りたいと思います。
「スマート・セル・インダストリー」について、その他、関連する資料としては、以下があります。
- 経済産業省 商務・サービスグループ 生物化学産業課 (平成29年12月19日)「スマートセルインダストリーの実現に向けた取組」
- 神戸大学・理化学研究所 バイオ小委員会「スマートセルインダストリーの基盤:合成バイオ技術の現状と今後望まれる開発」
2つの技術の相補関係
以上、2つの技術を見てきました。いま一度、整理すると、以下のようになります。
- CRISPR-Cas9:(自然界が生み出した)既存の塩基配列を編集することで、必要なゲノムを獲得する技術。
- 「合成生物学」(Synthetic biology):人間が白紙の状態から、「塩基」の文字でゲノム配列を書いていく方向性(アプローチ)を目指す技術。
須田『合成生物学の衝撃』は、この2つの技術は、互いに補い合う関係にあると指摘しています。同書の57-58ページ目を抜粋して転載します。
合成と編集のコンビネーション
「CRISPRは、合成生物学にとって信じられないほど役に立つ道具を提供した」。米ジョージ・メイソン大学で生物テロ防御などを研究し、合成生物学の進展に詳しいグレゴリー・コブレンツ教授はそう説明する。
「もし仮に合成生物学者がバイオブリックで車を作ろうとしているとしたら、それはハンマーとバールだけで作ろうとするようなものだ。彼らにはその2つの道具しかなかったのに、いまやドライバーとレンチを持っている。CRISPRは、より簡単で、かつ予測可能な方法で使える道具を彼らに提供したんだ。これによって、彼らのビジョンは実現に少し近づいた」
ちなみに、CRISPRを使って既存生物のゲノムを改変する場合でも、塩基配列を「合成」する技術は依然として必要だ。ターゲットとなる塩基配列を探し、ハサミ役の酵素を連れて行くガイド役のRNA、また新たに挿入する遺伝子のDNAは、化学合成によって作られる。
一方、ゲノム編集という使い勝手の良い道具が手に入ったからには、何も苦労して一から完全なDNAを作成する必要はないような気もする。ゲノムを必要な場所だけ改変すれば事足りるのではないか?だが、マサチューセッツ工科大学(MIT)と米国防総省の出費で設立されたリンカーン研究所の合成生物学者、ピーター・カーによれば、非常にたくさんの部位–たとえばゲノム全体で数万箇所–のゲノム改変を試みるときは、やはり一から合成していく方がずっと効率的でコストも抑えられるという。
また、カーは。「完全なDNAを合成する場合も、ゲノム編集は間違いを修復するために使われる」と指摘する。長いDNAほど作成過程でエラーが生じやすく、たった一文字の間違いが致命的な影響を及ぼすことがあるからだ。
こうしてみると、合成生物学の発展において、DNA/RNA合成とゲノム編集はどちらも欠かすことのできない相補的な技術と言える。
(MIT発)BioBrick: 機能単位の塩基配列ブロックを、積み木細工のように自在に組み合わせるアプローチ
MITのトム・ナイト氏とドリュー・エンディ氏は、塩基配列を機能ブロック単位に切り出して、レゴ・ブロックのように自在に組み合わせて、求めている機能を実現する人工生物部品をつくるBioBrickというアプローチを提案し、iGEM競技会の出場チームに対して、BioBrickブロックを無償で利用できるライブラリとして提供し、BioBrickライブラリが合成生物学の若手研究者と実務者の間で広く利用されるようになるように後押ししています。
以下、東洋経済オンラインの2018年5月6日付けの記事「最も勢いのある科学「合成生物学」の最前線 コンピュータ上で生命が設計されている」から、次の段落を引用して転載します。
合成生物学の大きな流れの一翼を担ってきたのは、トム・ナイトやドリュー・エンディといったMITの工学者たちである。生物学を「工学化」するーーそのようなコンセプトで彼らが夢見たのは、伝統的な生物学を掘り下げることではなく、トランジスタやシリコンチップに代えてDNA配列と細菌を用い、「生物マシン」を作るということであった。
特に工学的なアプローチとしての特徴が顕著なのは、バイオブリックという規格を作り出したことにある。後にそれはiGEMという、世界中から若い才能とアイデアが集まり、技術と課題を共有できる場へと発展した。
高校生向けの合成生物学の実験実習教育キット “BioBuilder”
合成生物学の講義は、MIT(マサチューセッツ工科大学)を始め、米国の複数の高等・中等教育機関で実施が始まっているようです。そして、高校生を対象に実施されたいくつかの実験的な授業で教壇に立ったに教諭からのフィードバックを受けて、学生・教諭の双方が、合成生物学の製作実習授業に取り組む上で手助けとなる教材「ガイドブック」として、BioBuilderというものが一般に刊行・販売されています。
日本語の翻訳書もすでに出版されており、紀伊國屋書店や丸善ジュンク堂などの大型書店のほか、アマゾンドットコムからオンラインで購入することができます。
また、以下の公式ウェブページからは、授業の様子を捉えたスナップ写真や、合成生物を組み立てるためのモジュール部品の購入画面への案内があります。
このように、実際に合成生物を製作する実験・実習を行うための環境が、いま急送に整えられ始めています。
BioBuilderの参考テキストが、O’Reilly社から刊行済み

『バイオビルダー ― 合成生物学をはじめよう』(O’Reilly Japan。 2018年11月21日刊行)

BioBuilderは、ソフトウェア工学におけるアジャイル開発技法や、ライブラリの標準化・共有利用化を合成生物学の領域に取り入れる先導役を目指している
このBioBuilderキットでは、合成生物学は、遺伝子工学(ゲノム工学)と合成生物学という生物学特有の学術理論とノウハウ・テクニックに基づきながらも、プログラミング言語によるソフトウェアのアジャイル開発現場や、機械工学の開発現場で長年にわたって培われてきた「部品のモジュール化」や「部品製品の仕様の標準化」を、合成生物学の世界に取り入れる必要があると、問題提起をしています。
「部品のモジュール化」と「部品製品の仕様の標準化」が、国際標準規格(統一規格)として策定されることで、世界中のエンジニアたちや研究者たちが、合成生物を「組み立てる」ために利用できる信頼性の高い「部品モジュール」を、安価に、かつすばやく入手(購入)することができるようになるような環境を整えなければならないと述べています。
そして、世界中のエンジニアや研究者が、標準化された部品モジュールを用いて「合成生物」を「組み立てる」ようになるようにすることを目指しているようです。
その結果、あるエンジニアや研究者が公開した「合成生物学」組成・構築の成果を、世界中の他のエンジニアや研究者が再現性テストを(同じ部品を用いて)行うことができるようになることで、合成生物学が、再現可能な科学(サイエンス)として、確立される日が到来する時期を早めることができるはずだ、と見ているようです。
高校生・大学生向けの合成生物製作・国際競技会 iGEM
合成生物学の知見と技法を用いて、実際に合成生物学を製作する動きを広めるために、高校生・大学生を対象にした国際的な技術競技会(コンテスト)が開かれています。
2004年の初開催以来、毎年開催されているiGEM(The International Genetically Engineered Machines)です。『バイオビルダー ― 合成生物学をはじめよう』(O’Reilly Japan。 2018年11月21日刊行)の16ページ目に掲載されたコラム欄によると、この競技会は、『「シンプルな生物システムを標準化された交換可能なパーツから構築し、生きている細胞中で機能させることができるかどうか?」』を競うもののようです。
iGEMの課題設定
iGEMに出場したチームは、それぞれ自由に、「合成生物」を用いてどのような課題の解決に取り組むのか、問題設定を登録することができます。
競技会では、設定した課題に対して、そのチームが(合成生物学の知見と技術を用いて)製作した「合成生物」が、その課題を解決しうる振る舞い(挙動)を、どれだけ正確に、安定して見せることができるのかが、採点されます。
課題の例として、『バイオビルダー ― 合成生物学をはじめよう』(O’Reilly Japan。 2018年11月21日刊行)では、水中に「ヒ素」が混入している場合、何らかの方法(例:細胞がの発行色が変わる、など)で周囲の人々に知らせることで、住人がヒ素が混じった水を誤飲してしまうのを防ぐという課題に取り組んだ過去の出場チームの事例が紹介されています。
iGEM出場チームが取り組んだ課題:”写真機として機能しうる人工生物資材”ほか
『バイオビルダー ― 合成生物学をはじめよう』(O’Reilly Japan。 2018年11月21日刊行)では、過去に開催れたiGEMの世界大会に出場した各大学のチームが、どのような「機能」(挙動)を発揮するアミノ酸構造体を作り出したのか、いくつか事例を紹介しています。
2004年出場(MITチーム):Eau d’coliプロジェクト。細胞分裂の進展段階の変化を「匂い」で(人間に)知らせるアミノ酸構造体を生み出す人工の塩基配列
このプロジェクトの成果は、MITのウェブページで閲覧することができます掲載。

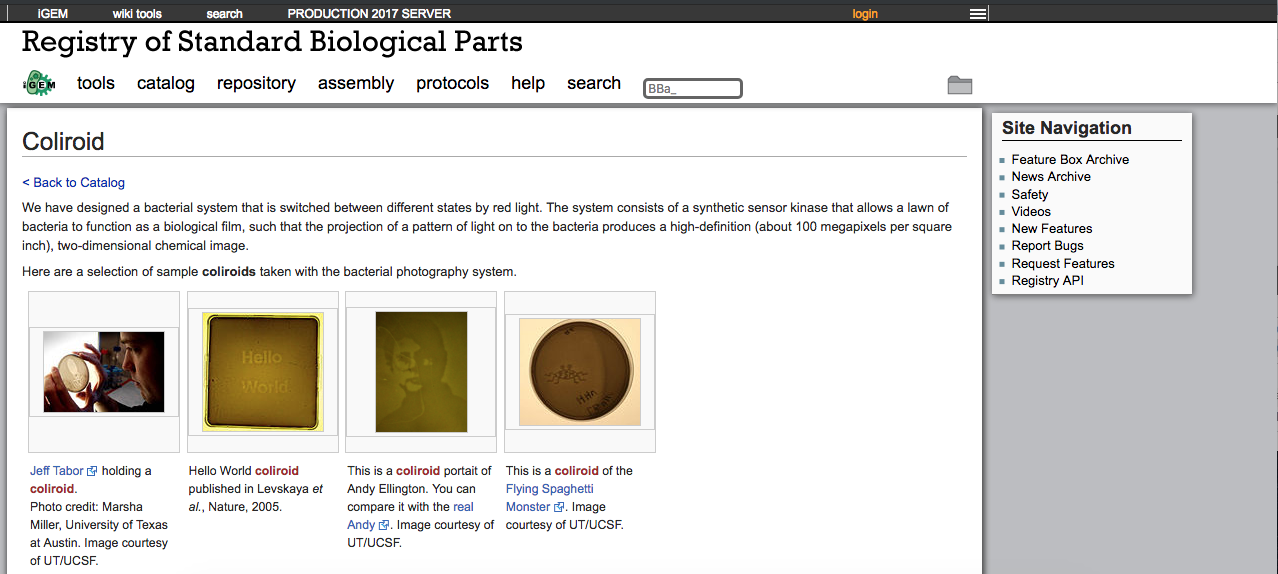
2004年出場(テキサス大学&カリフォルニア大学):Coliroidプロジェクト。「細菌写真器」(光検出器と発光器の組み合わせ)として機能する大腸菌塩基配列の組み換え
このチームが作り出したColiroidは、次に紹介する生体部品の登録ライブラリ”Registry“に登録されています。
以下が、そのウェブページです。
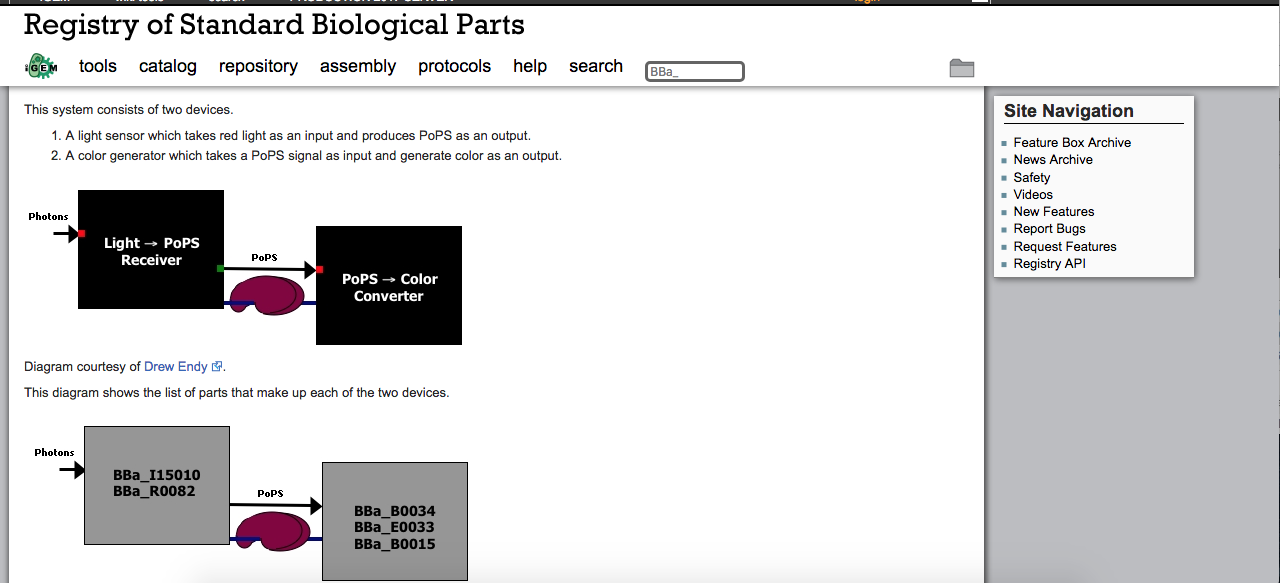
iGEM:Registryライブラリに登録された生体部品(BioBrick)を組み合わせてることで、電子部品や機械部品をつなぎ合わせる感覚で、人工生物資材を組成する
また、前述のO’Reilly社刊行の教本に掲載されているコラム欄には、以下の記述もあります。
各iGEMチームは、iGEMの「標準生物学的パーツ・レジストリ(Registry of Standard Biological Parts)」の中にある標準化されたパーツを使って、新規の生物システムの設計、構築に挑みます。
これらのパーツは標準化された接合部を持っていて、そのことによって一貫性のある再利用可能なアセンブリ方式で物理的にパーツを接続することができます。
チームは、わずか4つの制限酵素とiGEMライブラリにある標準化されたDNAパーツを使うことができ、遺伝子回路をアセンブリし、より複雑な遺伝子要素の配列を作ることができます。
標準生物学的パーツの再利用は、さまざまな学校のチームが試薬を共有し、夏季のプロジェクトの進捗を加速させる方法となっています。
Registry of Standard Biological Parts
Registry of Standard Biological PartsのURLは、http://parts.igem.org/Main_Pageです。以下がそのトップページの画面になります。
このRegistryのトップページにあるCatalogをクリックすると、生体部品(BioBrick)のカタログのウェブページ(http://parts.igem.org/Catalog)に飛びます。
Registry of Standard Biological Partsについては、http://igem.org/Registryに、全体像を説明する解説文を読むことができます。以下、冒頭部分を引用します。
The iGEM Registry of Standard Biological Parts is a growing collection of genetic parts that can be mixed and matched to build synthetic biology devices and systems. Parts submitted to the Registry through the iGEM competition follow the BioBricks assembly standard as described by RFC 10 and as such the vast majority of the parts in the Registry are RFC10-compatible.
As part of the synthetic biology community’s efforts to make biology easier to engineer, the Registry provides a source of genetic parts to iGEM teams during the annual competition and to academic labs through the Labs Program. Teams and other researchers are encouraged to submit their own biological parts to the Registry to help this resource stay current and grow year to year.
日本語版のWikipediaにも、“BioBrick”の項目が登録済みのようです。
BioBrick (バイオブリック) 標準生物学的パーツは定まった構造や機能を持つDNA配列のことである。これらは共通のインタフェースを持つように設計されており、新しい生物系を構成するために大腸菌などの生きた細胞に組み入れることができる。BioBrickパーツは合成生物学に工学原理の抽象化と標準化を取り入れるために作られた。
BioBrickパーツはマサチューセッツ工科大学のTom Knightにより紹介された。[1]スタンフォード大学のDrew Endy[2] とカリフォルニア大学サンフランシスコ校のChristopher Voigtもこのプロジェクトに深く関わっている。公開された数千ものBioBrickパーツの登録はRandy Rettbergらが管理している。[3] 大学生や大学院生が生物学的デバイスを構築して競い合う大会であるiGEMには、BioBrickパーツの普及を推進する狙いがある。
BioBrickパーツには「パーツ」「デバイス」「システム」の3種類がある。[2] 「パーツ」はブロックを構成したり基本的な生物的機能を持つもの (例として、タンパク質をコードするもの、プロモーターを提供してRNAポリメラーゼを下流配列に結合させて転写開始させるもの)、「デバイス」は定義された機能を果たすパーツの集合体 (例として、特定の化学物質が存在する際に蛍光タンパクを生産するもの)、「システム」は高度な機能を果たすもの (例として、2つの色が周期振動するもの) である。
これまでiGEMで作製されたBioBrickシステムの例としては以下のようなものがある。
生物学的パーツの性能を測る2つの尺度はDrew Endyらによって定義された。1つはPoPS (Polymerase per second) と呼ばれる、RNAポリメラーゼが一定のDNAの点を1秒間に通過する数、もう1つはRips (Ribosomal initiations per second) と呼ばれる、リボソームが一定のmRNAの点を1秒間に通過する数である。[4]
上記の説明文のなかに、iGEMに出場した学生たちが作成したBioBrickが、BioBrickの共有資産に追加登録されていることを述べた一文を確認することができます。
BioBrickの生体部品の取扱説明書(技術仕様書)の例
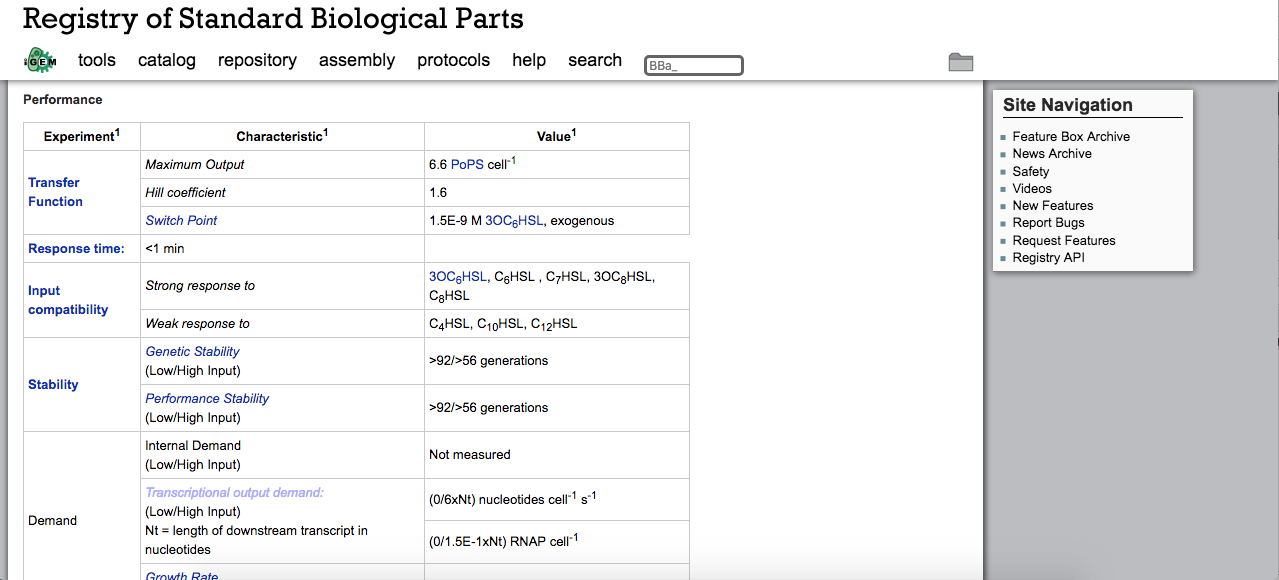
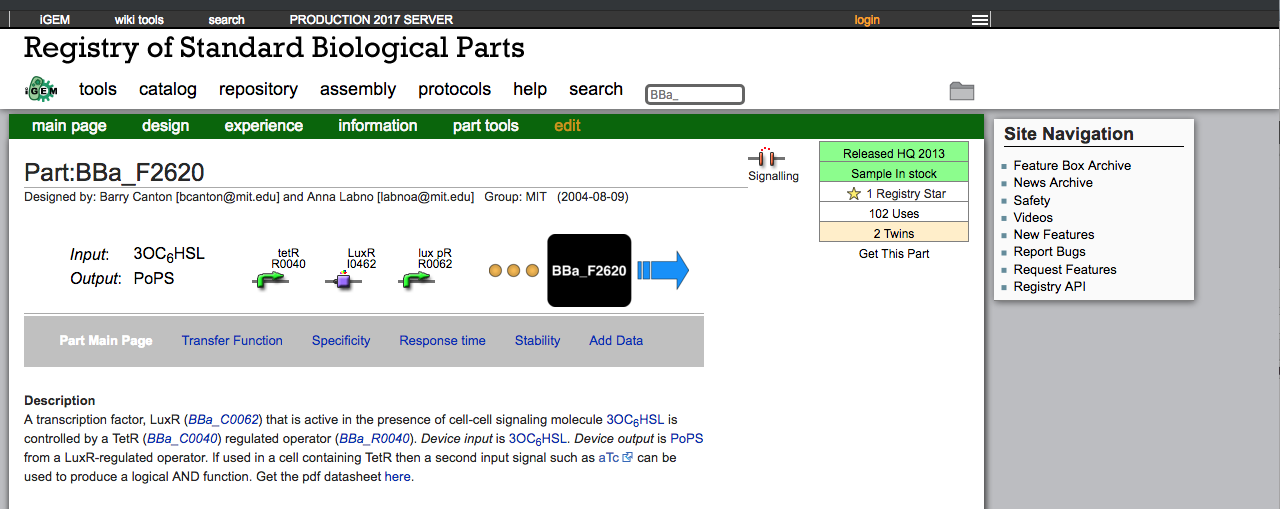
『バイオビルダー ― 合成生物学をはじめよう』(O’Reilly Japan。 2018年11月21日刊行)の132ページ目には、BioBrickの生体部品の取扱説明書(技術仕様書)の一例として、BBa_F2620というコードネームでRegistryに登録された生体部分(BioBrick)の仕様書を掲載しています。
ここでは、この「生体部分(BioBrick)」が、入力情報として、どのような生体信号を受け取って、どのような生体信号を出力するのかが、定義されています。
また、入力信号の強弱や属性が動的に変化した場合、このBioBrickから出力される生物信号が経時的(時系列的)にみて、どのように変化するのか(電子工学や機械工学における制御工学の入出力応答関数)も、部品の「仕様書」として、測定値が記載されています。
部品となる塩基配列
BioBrickとして、主なものは、以下のものがあります。以下のようなBioBrickを記述するDNAの塩基配列を、単語を組み合わせて文を綴るように、しかるべき文法規則(ゲノム言語の規則)にのっとって、適切な順序で記述していくのが、合成生物学における「設計」の作業になります。
- DNAを転写してmRNAを生み出す開始信号である「プロモーター」を記述する塩基配列
- mRNAの設計情報をタンパク質に翻訳するリボソームを招く「RBS」(リボソーム結合部位)を記述する塩基配列
- 塩基配列上、後方に記述された塩基配列(プロモーターやRBSなど)の働きを促進させたり、抑制させる因子を記述する塩基配列(「遺伝子発現レギュレーター」など)
なお、この記事でも触れた理化学研究所による人工の塩基配列の創出は、「ゲノム言語の規則」にのっとって、利用することのできる新たな「文字」を、人類が自然界に追加したことを意味します。
出力を反転させる「NOT論理ゲート」や「AND論理ゲート」・「OR論理ゲート」も登録済み
『バイオビルダー ― 合成生物学をはじめよう』(O’Reilly Japan。 2018年11月21日刊行)の105ページでは、自然界にすでに存在する生体資材として、NOT論理ゲートやAND論理ゲートやOR論理ゲートなど、計算機(コンピュータ)のCPUチップに搭載されている論理回路に相当する機能(挙動)を発揮するような資材が存在することが紹介されています。このような「論理回路」として用いることができる資材も、Registryに登録済みです。
連続値を入出力する「アナログ」部品である生物試料を組み合わせることで、オン・オフの非連続な「デジタル」入出力に近い挙動を実現する手法も開拓済み
また、同書の155ページでは、以下の段落があります。そのまま引用します。
ほとんどの自然システムはアナログであり、入力信号のわずかな変化でアウトプットがより段階的に増加します。合成生物学者は、いくつかの賢明な手法を使って、よりデジタルな振る舞いをする細胞を実現しています。例えば、アナログ制御を複数の層で構築して信号を重ねることで、アウトプットがあたかもオンあるいはオフのどちらかに見えるようにすることです。
BioBricksを連結させることで、アルゴリズムを実行可能なバイオ論理回路を組むことも可能か
BioBricksを連結させることで、アルゴリズムを実行可能なバイオ論理回路を組むことも可能であるのではないでしょうか?
DNAがもつ化学的な振る舞いの特性を活用することで、論理回路を設計する研究は、さかんに行われています。
山椒魚 (2016/06/30)「DNAでフリップフロップ回路を設計してみた DNA based flip-flop」より転載。
山椒魚 (2016/06/30)「DNAでフリップフロップ回路を設計してみた DNA based flip-flop」より転載。
- Wired(日本語版)2017.06.24 SAT 19:00 『ヒトの細胞がプログラミング可能に:米研究チームが109通りの「論理回路」の作製に成功』
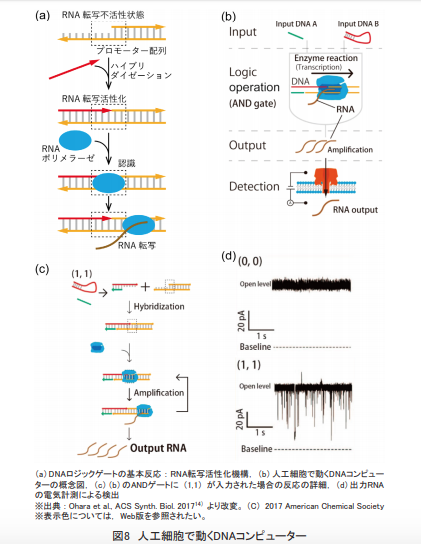
- [東京工業大学] 東工大ニュース(2017年5月16日付け)「人工細胞の中でDNAをコンピュータとして使うことに成功―生体内で働く分子ロボットの実現に向けて―」
また、DNA以外にも、自然界にある分子の化学的な挙動の特性を活用して、論理ゲートを構築する研究が行われています。
UCLAとHP社の研究チームは、コンピューターの動作の基本を形作る、分子的「論理ゲート」をつくり出した。
「コンピューターで使用される論理ゲートの、最も単純な形のものを実際に作ることに成功し、それは稼働した」とカリフォルニア州パロアルトにあるHP社のコンピューター設計者、フィル・キュークス氏は電話インタビューで述べた。
論理ゲートとは、情報のビットの状態を示す「オン」と「オフ」を切り替えるもので、その切り替えは、電圧の切り替えによって行なわれる。
ヒース氏の研究チームは、『ロタクサン』(rotaxane)と呼ばれる新しい化合物を開発することでこの論理ゲートを成功させた。この化合物は結晶構造で成長する。
ヒース氏およびキュークス氏の研究チームが『サイエンス』誌に寄せた内容によれば、ロタクサン分子を金属の電極で挟むと、論理ゲートとして機能するという。
なお、以下のような研究の取り組みも進んでいるようです。生体分子を用いた論理回路(biological circuits )を用いた演算アルゴリズムを、専用のプログラミング言語を用いることで、ソース・コードとして記述できることに道を開こうとする試みです。
- Munenori Taniguchi (2016/04/15) 『DNA用の「プログラミング言語」をMITが開発。望みの機能をコーディング~コンパイルし、細胞へインストール』
- Alec A. K. Nielsen et.al., Genetic circuit design automation, Science, 01 Apr 2016:Vol. 352, Issue 6281, aac7341
RESULTS
Cello designs circuits by drawing upon a library of Boolean logic gates. Here, the gate technology consists of NOT/NOR logic based on repressors. Gate connection is simplified by defining the input and output signals as RNA polymerase (RNAP) fluxes. We found that the gates need to be insulated from their genetic context to function reliably in the context of different circuits. Each gate is isolated using strong terminators to block RNAP leakage, and input interchangeability is improved using ribozymes and promoter spacers. These parts are varied for each gate to avoid breakage due to recombination. Measuring the load of each gate and incorporating this into the optimization algorithms further reduces evolutionary pressure.
Cello was applied to the design of 60 circuits for Escherichia coli, where the circuit function was specified using Verilog code and transformed to a DNA sequence. The DNA sequences were built as specified with no additional tuning, requiring 880,000 base pairs of DNA assembly. Of these, 45 circuits performed correctly in every output state (up to 10 regulators and 55 parts). Across all circuits, 92% of the 412 output states functioned as predicted.
CONCLUSION
Our work constitutes a hardware description language for programming living cells. This required the co-development of design algorithms with gates that are sufficiently simple and robust to be connected by automated algorithms. We demonstrate that engineering principles can be applied to identify and suppress errors that complicate the compositions of larger systems. This approach leads to highly repetitive and modular genetics, in stark contrast to the encoding of natural regulatory networks. The use of a hardware-independent language and the creation of additional UCFs will allow a single design to be transformed into DNA for different organisms, genetic endpoints, operating conditions, and gate technologies.
米マサチューセッツ工科大学(MIT)の生物工学者を中心とする研究チームが、生細胞に新たな機能を与える、DNAにエンコードされる複雑な回路を迅速に設計できるプログラミング言語を開発した。
この言語を使うと、特定の環境条件の検知やそれに対する反応を定義するプログラムを誰でも記述できるようになり、そのプログラムから機能を実現するDNAシーケンスが生成できるという。
「これは文字通りバクテリアのためのプログラミング言語だ」と、MIT生物工学教授のクリストファー・ヴォイト(Christopher Voigt)氏は述べる。「コンピューターのプログラミングと同様に、テキストベースの言語を使用する。そのテキストをコンパイル(実行できるコードに翻訳)してDNAシーケンスに変換し、細胞に組み込めば、細胞内で回路が機能する」。
Science誌に4月1日付で発表された研究論文によると、ヴォイト氏とボストン大学および米国立標準技術研究所の研究チームは、この言語を使って、3種類の入力を検出して異なる方法で応答できる回路を作成した。将来的な応用としては、腫瘍を検出したら制癌剤を生産するバクテリアの設計や、有毒副産物が過多の場合に発酵プロセスを停止できる酵母細胞の作成などが考えられるという。
既存のCPUでは実現が難しいことがバイオ論理回路で可能になるか?
ところで、分子の化学反応を活用した演算は、電子の動きで演算を行う既存のCPUやNeuromorphic computerチップよりも、計算処理の速度が遅いはずです。
しかし、バイオ生体部品(BioBricks)は自己増殖性機能(細胞分裂)や自己修復機能をもつことが期待されます。
CPUや、現在世界中で開発中のNeuromorphic computerチップでは、実現が難しいと考えれることを実現できるかもしれない、技術的な可能性を秘めています。
(参考)Neuromorphic computer chipについて
ところで、汎用的な計算タスクを行うことができるCPUとは別に、深層ニューラル・ネットワークモデル(ディープ・ラーニングモデル)の演算処理を行うことに特化した(逆にいうと、深層NN以外の演算処理を行うことは、回路構造のつくりから、できない)論理回路を(物理的に)設計することを目指すNeuromorphic computerチップが、IBMなど各社によって、研究開発が進んでいます。
「DNAコンピュータ」という用語は、別の原理で動くコンピュータの技術構想を指す
なお、「DNAコンピュータ」という言葉が一般に指すのは、塩基配列を組み合わせて論理回路を構成する計算機ではありません。
一般には、「DNAコンピュータ」というと、1994年に、米国・南カリフォルニア大学のレオナルド・エイドルマン教授が概念を提唱した計算機の機構のことを指します。これは、AがT、GがCと結合する塩基どうしの化学的な性質を活用することで、巡回セールスマン問題の解を並列処理で求めるものを指します。
- ■DNAコンピュータ- エイドルマンの発想
- ■DNAコンピュータ - いろいろな酵素をつかった複雑なDNAコンピュータ
- ■DNAコンピュータ - DNAの基礎知識
- ネイチャーインタフェイス「DNAコンピューター 遺伝子を利用した超高速コンピュータ誕生」
- Wired誌(日本語版)2003.08.26 TUE 05:00配信「着想から10年、DNAコンピューターの現状と課題」
「生体分子ロボット」研究の試み
生体分子を用いて、頭脳系(知的情報処理)と感覚・運動系を担う生体分子ロボを作る試みも行われています。
人間の身体や脳のなかで、このような分子サイズの知的情報処理(分類タスクや予測タスクや強化学習タスクをこなせる機械学習モデル)と、感覚・運動系を備えた「分子ロボット」が常時稼働することで、個々人の健康管理や能力増強が行なわれるようになる世界は、まさに、カーツワイル氏がGNR論のなかで展望している近未来の光景です。
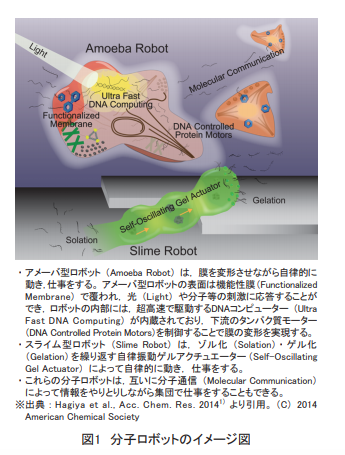
2.3 分子ロボティクスプロジェクト「感覚と知能を備えた分子ロボットの創成」の試み
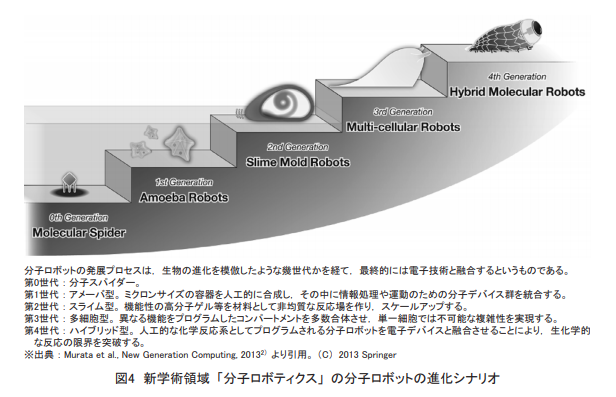
近年,このような分子ロボティクス分野において,ナノサイズのロボットを超え,細胞のように「感覚」や「知能」といった複雑な統合機能を備えた,単独で自律的に駆動する「統合分子ロボット」を構築していこうという試みがなされている。
特に,新学術領域「分子ロボティクス」(http://www.molecularrobotics.org/)は,日本での本分野の開拓と発展に大きく貢献した(図4)。
このプロジェクトでは,感覚を構築する班,知能を構築する班,感覚・知能の機能を統合して,動きにつなげる2つの班(ミクロサイズのアメーバ型,マクロサイズのスライム型)によって構成されていた。
感覚を構築する班では,DNA/RNA/タンパク質等を用いて分子センサーデバイスを構築し,感知した分子情報を,分子ロボット内部へ分子的に伝達する技術を構築した。
知能を構築する班では,DNAコンピューティング技術を基盤として,自律的に分子的に計算し,結果を出力することで下流を制御できる知能デバイスを研究した。
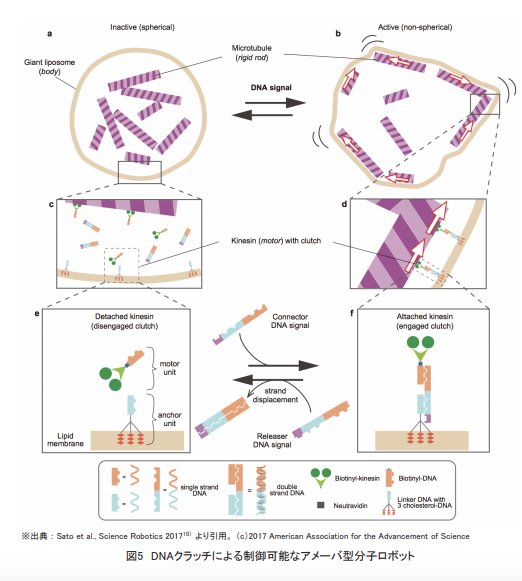
それらの技術を統合する形で,アメーバ型やスライム型の分子ロボットの構築を行った。 感覚と知能を備えた,アメーバ型分子ロボットの実証このプロジェクトを通し,アメーバ型の分子ロボットの構築で大きな成果が得られ,2017年にロボット工学のトップジャーナルであるScience Robotics誌(米国・Science誌の姉妹誌)に論文が掲載された10)(図5)。
この研究には,東北大学の野村准教授の研究グループが中心的な役割を果たした。
瀧ノ上 正浩 「生体内で働く分子ロボットの実現へ 情報媒体としてのDNA分子とDNAコンピューティング」情報管理 2017.12. vol. 60 no. 9より転載。
瀧ノ上 正浩 「生体内で働く分子ロボットの実現へ 情報媒体としてのDNA分子とDNAコンピューティング」情報管理 2017.12. vol. 60 no. 9より転載。
瀧ノ上 正浩 「生体内で働く分子ロボットの実現へ 情報媒体としてのDNA分子とDNAコンピューティング」情報管理 2017.12. vol. 60 no. 9より転載。
瀧ノ上 正浩 「生体内で働く分子ロボットの実現へ 情報媒体としてのDNA分子とDNAコンピューティング」情報管理 2017.12. vol. 60 no. 9より転載。
研究者が所望する特性を持った生命システムを、設計し“つくる”研究が近年行われています。生体分子による化学反応の自律制御を利用した計算機をつくる“分子コンピューティング”、生体分子から成る構造物やアクチュエータをつくる“分子ロボティクス”、人工の生体分子ネットワークを組み込むことにより所望の特性を生物に持たせる“合成生物学”を当研究室では研究しております。これらの研究には、生命システムの原理の理解という理学目的や、分子ロボットや細胞をシャーシとした生物ロボットを医学・工学的に役立てる目的があります。
分子コンピューティング: DNAは、A、C、G、Tいずれかの塩基を含む4種のデオキシヌクレオチドが連なった直鎖状分子の総称です。このDNAを4進数の文字列とみなし、遺伝子工学的手法でDNAを切ったりつなげたりして文字列を変換することで計算を行います。 <詳しくはこちら>
分子ロボティクス: DNAを素材とした一般的な二本鎖とは異なる任意の形の構造物、DNAの鎖置換反応を利用したアクチュエータ、特定の小分子に応用するRNAなど生体分子から成るパーツを開発することが可能ですし、それらを組み合わせて分子ロボットを作製することができます。 <詳しくはこちら>
合成生物学: 他の生物の遺伝子や改良した遺伝子を細胞に導入することで、様々な特性を生物に与えることが出来ます。この10年でトグルスイッチやオシレータ、論理ゲートなど電子部品を模した多様な機能を細胞に持たせることに成功しています。これら“遺伝子回路”の開発には数理モデルをベースとした工学的手法による設計が不可欠であり、生物学者・工学者、様々な人材が合成生物学の分野に参入しています。当研究室では遺伝子回路の設計や解析といった計算機による“DRY実験”や実際に遺伝子回路を開発し生物に導入する“WET実験”を行うことが可能です。 <研究例>
このように、「生命をつくる」研究とは、生命の部品あるいは生命自体を素材とした“ものづくり”の新しい分野です。 世界中で腕に覚えのある人たちが、アイデアで競い合っているような楽しい学問です。
BioBrickを連結させた場合の挙動をシミュレートするCADや計算機シミュレータの開発が目指されている
『バイオビルダー ― 合成生物学をはじめよう』(O’Reilly Japan。 2018年11月21日刊行)では、電子工学や機械工学において、制御工学の計算機シミュレーションやコンピュータ支援設計(CAD: Computer Aided Design)が広く行われているように、合成生物学においても、実際にBioBrickを実験台の上でつなぎあわせる作業に入る前に、パソコン上で、目指している人工生物資材を生み出すために、どのBioBrickとどのBioBrickを、どのような配列順序や条件のもとで、つなぎあわせればよいのかをシミュレーションすることができる環境を整備するための努力が進行中であると報じています。
Standard Biological Parts Knowledge baseデータセット
なお、生体部品を結合させる合成生物学(Synthetic biology)の研究者とエンジニアのために、生体部品(BioBrick)を、データベース検索言語RDFなどを用いて論理検索を行うことができるSemantioc Web形式で収集・登録したものとして、SBPkb: Knowledgebase of Standard Biological Partsがあります。
論文としては、以下があります。
iGEM 2018年大会
以下は、2018年度の国際大会の公式ウェブページです。
日本からは、東京工業大学がこのiGEMに複数年、チームが出場しており、金賞を11年連続で獲得しつづけていることが、同大学の広報ウェブページに掲載されています。

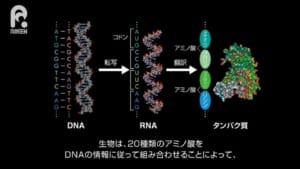
合成生物学の目的
合成生物学は、以下の3つの目的をもっていると整理することができるのではないでしょうか。
- 人体の強化:病原菌やウイルスに対する耐性の付与、過酷な自然環境や生活条件で健康に長期間、活動できる能力の付与など
- 産業上、有用なエネルギーや物質を生産する生物細胞の創造、それ自体有用な生物資源の創造:スマート・セル・インダストリー
- 生命の仕組みを「作ることで理解する」構成論的科学:AI✕ロボティクスで実行されてきた手法を、実際に塩基やアミノ酸やタンパク質を操作して、生物の有機細胞を用いて行う
3の「作ることで(人間を始めとする生命の活動を)理解する」という「構成論的科学」は、これまで、コンピュータ科学と人工知能科学の領域で行われてきました。
そこでは、計算論的(脳)神経科学などの知見をもとに、人間の知性を模倣した知能モデル(人工知能モデル)を計算機上に展開して、シミュレーションを行うことで、人間の知能(知性)の原理を理解しようとする取り組みが、行われています。
また、東京大学の國吉 康夫・新山 龍馬研究室や大阪大学の浅田稔研究室は、母体の子宮のなかで生育している段階にあるヒトの胎児の四肢(腕と脚)の関節や筋肉や神経系などの物理的な構造を、数理モデルとして記述することで、最初は無秩序に自分の手足を動かしている胎児が、どのような力学的なメカニズムを通じて、どのようなプロセス(経過)を辿ることで、みずからの身体の使い方(身体図式)をじょじょに獲得していくのかを、計算機シミュレーションを行うことで、解明する取り組みが行われてきました。これも、計算機上で数理モデルを「作ることで(胎児における身体図式の認識過程と、神経・筋肉体系の発育過程を)理解する」構成論的アプローチのひとつです。
今後は、こうした数理モデルを用いた「作ることで理解する」取り組みは、現実に対象としている生物と同じ、塩基やアミノ酸やタンパク質を用いて行うことが、一般化するかもしれません。
また、「作ることで理解する」科学では、自然界にコレまで存在しないが、原理的にはこの地球上で成立しうるあらたな「人工生命体」の可能性を模索する研究も、計算機シミュレーション上で取り組まれてきました。
今後は、合成生物学の知見と技術を用いることで、実際にタンパク質の「身体」をもった「人工生命体」が作られる時代が到来しつつあることが、予想されます。
(レベル3) 人間が、ゲノム言語に新たな「文字」を追加する技術
ゲノム言語に、自然界に存在しなかった新しい2つの塩基(DsとPx)を追加した日本の理化学研究所
3番目の技術については、我が国の理化学研究所が、「Ds」という塩基と、「Px」という塩基を、人工的の新たに生み出すことに成功しています。

今回、理研ゲノム科学総合研究センター タンパク質構造・機能研究グループと国 立大学法人東京大学は共同で、自然界に存在する塩基対と同じように遺伝的複製と転 写を繰り返す機能を持つ「人工塩基対”Ds-Pa(ディーエスピーエー)”」の開発に成 功しました。
これは、産業応用に欠かせない「複製」が機能するという世界で初めて の人工塩基対になります。
以下、上記のプレスリリースの一部を抜粋して転載します。
理化学研究所(理研、野依良治理事長)と理研ベンチャー[1]のタグシクス・バイオ(平尾一郎代表取締役)は、自然界には無い人工塩基を天然のDNA[2]に組み込むことで、DNAの機能を飛躍的に向上できることを世界で初めて証明しました。これは、仮説が提唱されてから約50年後の実証で、次世代の遺伝子操作技術にも大きく貢献するものであり、理研生命分子システム基盤研究領域(横山茂之領域長、現:横山構造生物学研究室 上席研究員)核酸合成生物学研究チームの平尾一郎チームリーダー(現:ライフサイエンス技術基盤研究センター[渡辺 恭良センター長] 構造・合成生物学部門 生命分子制御研究グループ 合成分子生物学研究チーム チームリーダー)らによる共同研究グループの成果です。
遺伝子の本体であるDNAは、糖とリン酸と塩基で構成されている核酸です。4種類の塩基(A、G、C、T)からなる複製[3]可能な情報分子ですが、抗体(DNAアプタマー[4])や酵素としての機能も兼ね備えています。しかし、わずか4種類の塩基からなる核酸の機能には限界があります。こうした中で「塩基の種類を人工的に増やすことができれば、核酸の機能が飛躍的に向上するのではないか」という仮説が、1962年に提唱されており、幾つかの研究チームが先駆けて新たな塩基(人工塩基)の開発競争を進めていました。
共同研究グループは、2009年に複製可能な人工塩基対「Ds-Px(ディーエス-ピーエックス)」を世界で初めて開発しました。さらに今回は、“人工塩基Ds”をDNAに組み込み、標的のタンパク質だけに結合する「DNAアプタマー」の作製に成功しました。このDNAアプタマー中には2~3個のDsが含まれているだけですが、天然型塩基だけで構成される従来のDNAアプタマーと比較して、標的タンパク質との結合能力が100倍以上も向上しました。これにより、塩基の種類を増やすとDNAの機能が向上するという仮説を世界で初めて証明しました。この成果は、DNAなどの核酸を利用した診断技術や医薬品を開発する新たなバイオ技術を提供し、人工塩基対による次世代の遺伝子操作技術への道をひらきます。
以下は、上記の理化学研究所の研究成果を紹介したYouTube動画「Technology: powering the future~合成生物学編」の一部の画面キャプチャです。
この動画のなかでは、実際に研究チームを率いている研究者が出演して、この試みの概要と、将来目指していることなどが、一般の視聴者向けにわかりやすくかみくだいだ言葉で説明されています。


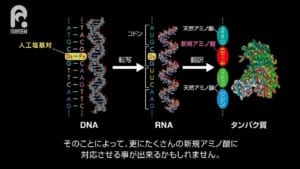


自然界にこれまで存在しなかった新たな「塩基」の出現によって、可能になること
先ほど引用した2本目のプレスリリースでは、以下の記述がありました。
「塩基の種類を人工的に増やすことができれば、核酸の機能が飛躍的に向上するのではないか」という仮説が、1962年に提唱されており、幾つかの研究チームが先駆けて新たな塩基(人工塩基)の開発競争を進めていました。
塩基対の法則とDNAの構造は、1953年にジェームズ ワトソンとフランシス クリックによって発見されました。それから間もない1962年には、タンパク質合成に関わるtRNAの立体構造を初めて明らかにした米国のアレキサンダー リッチが、「もしDNAの塩基の種類を増やすことができれば、DNAの情報や機能を拡張できる可能性がある」という仮説を提唱しました。
この可能性を証明するには、塩基の種類を増やしたDNAが正確に複製されるように第3の塩基対を人工的に作り出す必要があります。
そして、1990年以降、幾つかの研究チームが先駆けて第3の塩基対(人工塩基対)の開発競争を始めました。
この開発競争の中で、平尾チームリーダーらは2002年に最初の人工塩基対を開発し(Nature Biotechnology, 20, 177-182, 2002)、2006年には世界初の複製で機能する人工塩基対の作製に成功しました(Nature Methods, 3, 729-735, 2006、2006年8月24日プレスリリース)。
さらに、2009年には試験管内でDNAを複製させるPCR[5]という手法で、天然型塩基対に近い精度で複製するDs-Px(ディーエス-ピーエックス)塩基対を作製しました(図1)。
こうして6種類の塩基から成る複製可能なDNAを開発し、その後は、アレキサンダー リッチの提唱する「塩基の種類が増えるとDNAの機能は向上するのか?」が具体的な研究課題になりました。
上記で言及されているAlexander Rich氏の1962年の研究論文は、以下です。
なお、Wikipedia日本語版によると、このAlexander Rich氏は、以下のように分子生物学と遺伝子工学において大きな足跡を残した研究者であるようです。
アレクサンダー・リッチ(Alexander Rich, 1925年11月15日 – 2015年4月27日)は アメリカ出身の生物学者・生物物理学者。マサチューセッツ工科大学、ハーバード大学医学部の教授を務める。 コネチカット州ハートフォード生まれ。
ハーバード大学医学部卒業後、カリフォルニア工科大学のライナス・ポーリングのもとで博士研究員として働き、1958年マサチューセッツ工科大学に移った。
通常DNAの二重らせんは右巻きであるが(B-DNA)、1979年左巻きのDNAを発見しZ-DNAと名付けた。また転移RNAの立体構造を解明した。全米科学アカデミー会員。2015年ボストンで死去[1]。
平尾チームリーダーの論文としては、以下があります。
- 平尾 一郎 & 木本 路子 「遺伝情報を拡張する人工塩基対技術から新たな研究領域Xenobiologyに向けて」, Kagaku to Seibutsu 54(11): 835-840 (2016)
- (同 PDF版)
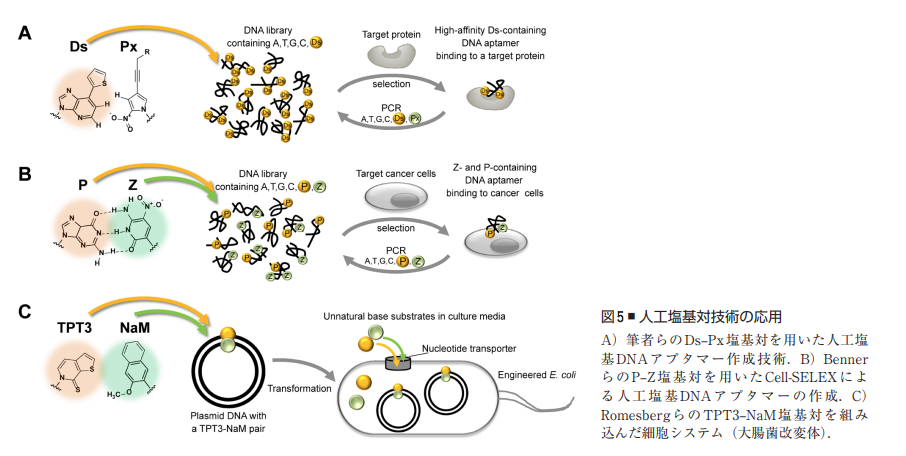
平尾 一郎 & 木本 路子 「遺伝情報を拡張する人工塩基対技術から新たな研究領域Xenobiologyに向けて」所収の「図5 ■ 人工塩基対技術の応用」を転載
2.人工知能・機械学習技術は、遺伝子工学・合成生物学の技術発展にどう関わっているのか?
標的以外の箇所の塩基配列を誤って編集してしまうエラーを予測する機械学習モデルの登場
遺伝子工学や合成生物学に対して、数理統計学・機械学習(人工知能技術)の手法を適用する事例としては。Microsoft社のCRISPR.MLライブラリが、その先駆的な試みとして挙げることができます。
30億文字の塩基配列が(複数の染色体にまたがって)並んでいる「長文」の遺伝子「文書」のうち、狙っている特定の1文字もしくは数文字(〜数十・数百文字)の塩基配列だけを標的(ターゲット)にして、意図したとおりに文字を削除したり、書き換えたりする際に、狙った文字列(塩基配列)とは異なる別の箇所(領域)の塩基配列を、間違って削除したり、書き換えてしまうエラーが発生することがあります。
この誤りエラーのことを、Off-targetと呼びます。
Microsoft社がリリースしたこのCRISPR.MLライブラリは、機械学習アルゴリズムを用いることで、このOff-targetと呼びれる「誤りエラー」が発生する確率を、事前に予測することができます。これにより、遺伝子書き換えをおこなう上で、Off-targetがなるべく起きないような方法を検討することが可能になると期待されています。
Microsoft発:CRISPR.ML ライブラリ
このライブラリを報じている記事としては、GIZMODOの2018年1月14日付けの記事「遺伝子編集技術CRISPRのエラーが機械学習で予測できるように」があります。
以下、この記事の冒頭部分を引用します。
オフターゲット効果とは、編集する予定ではなかったDNAにも影響を与えてしまうというもの。その結果、意図せずに癌などの新しい問題を生み出してしまうことがあり得るようです。オフターゲット効果の頻度がどれくらいか、というのはケースによるものの、正確な遺伝子編集技術といわれるCRISPRとはいえ、まだまだ完璧とは言えないのが現状なんですね。
こういった事故を減らすために、科学者たちは長年CRISPRの精度を高める方法を研究してきました。そんな中、Microsoftは人工知能がこの問題を解決してくれるのでは、と考えているようです。
Microsoftは米国中の研究施設のコンピューター科学者、生物学者らと協力して、Elevationと呼ばれる新しいツールを開発しました。これはCRISPRのオフターゲット効果を予測するというもの。
Microsoft社のこのライブラリについては、AINOWの過去の記事の中の以下の行の文章でも、以下の形で言及がなされています。
彼と彼の同僚が、ゲノム編集における最善の実験方法を決定するためにAIを活用して生物学的演算を実行するツールであるCRISPR.ML※を組み立てている時、機械学習のモデルを選択する作業は大きな負担となり、限界に達した。
上の記事では、上記の文章似続けて、さらに引用符を付して以下の文章を引用しています。
※CRISPR MLとは、ゲノム編集を効率的に実行するために開発された機械学習システム。遺伝子を改変する最新技術であるゲノム編集においては、改変すべきを遺伝子をガイドRNAと呼ばれる核酸(化学物質)で特定する。このガイドRNAが特定すべき改変に関係している遺伝子部位には、多数の候補があることが一般的である。同機械学習は、こうした遺伝子の候補から改変を実行するのに有効な遺伝子を特定することを目的に開発された。
“Off-target effects are something that one would really want to avoid as you want to make sure that your experiment doesn’t mess up something else,” said Nicolo Fusi, a researcher at Microsoft’s research lab in Cambridge, Massachusetts. Off-target scores: Elevation for every guide provides researchers with two kinds of Off-target scores: Individual scores for one target region. Single overall summary score for that guide. Target scores are probabilities that are based on machine-learning and are provided for every single region of the genome that something negative could happen. For every single guide, Elevation returns hundreds to thousands of these off-target scores.
iGEM出場チームによる機械学習・深層学習モデルの活用事例
iGEM出場チームのなかには、設定した課題を解決する過程で、深層学習(ディープ・ラーニング)モデルを含む機械学習モデルを用いたデータ解析を行ったチームがあります。
これは、合成生物学におけるAI(人工知能)の活用事例とみなすことができます。
以下に、iGEMに出場するにあたり、機械学習を用いたデータ解析を行ったチームを、2件、紹介したいと思います。
[ 事例1 ] scikit-learnライブラリのMLP(多層パーセプトロン)を用いた事例
- 台湾チーム:http://2017.igem.org/Team:CCU_Taiwan/Machine_learning
Introduction
We use machine learning to generate a model in order to predict user’s risk of caries. We took ICDAS as our standard data and train our model with CSP, lactate, concentration and pH value. In the following section, we will introduce how we preprocess our data for training the model and how we generate the model using scikit learn.
( 中略 )
Model generation
We use scikit-learn as a tool for generating the machine learning model. We choose the neural network MLP Classifier to train the prediction model. Although the accuracy of the prediction model at first isn’t that accurate due to the lack of data, but we look forward to collecting more data in order to improve the accuracy of the model.
iGEMに出場した台湾チームのウェブページに掲載されている図表を転載(http://2017.igem.org/Team:CCU_Taiwan/Machine_learning)
[ 事例2 ] R言語のSVM(サポート・ベクター・マシン)と全結合ニューラル・ネットワークモデルを用いた事例
- エジプトチーム:http://2018.igem.org/Team:AFCM-Egypt/Machine_Learning
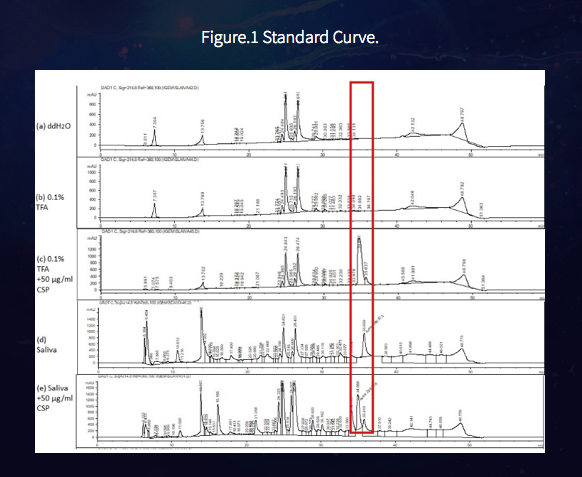
The aim of this deep learning is to provide an efficient classifier for predicting the likelihood of TLR binding of various DNA oligonucleotides through sequence features of binding motifs and CpG content.Analysis Summary( 中略 )ODN classification modelWe have categorized ODN into binders or non-binders according to Vaccine SVM algorithm. Hence, the classification predicts which ODN will bind to TLR or not based on testing through A deep feedforward fully connected ANN.R packages for deep learningWe have installed essential R packages for deep learning including, TensorFlow, K Keras, tidyverse and DNAshapeR.Network model definitionsWe have implemented the sequential model through Keras which includes a dense layer which defines a standard neural network with proper connections between input (ODN vector) and output (binding classification) followed by a dropout layer which helps to prevent overfitting the model.( 中略 )ConclusionsIn this model we have provided a straightforward example of deep learning usage in classifying DNA oligos on the basis of TLR binding which could facilitate studies of metagenomic DNA effects on innate immunity and inflammatory pathways of various human diseases.Although the accuracy of prediction have gone up for approximately 95% of accuracy, we still need to develop better deep learning models to avoid the over fluctuations of complex deep learning models during the process of parameter adjustment, we also need to train our models on curated datasets of proved significant as this model could be considered a model training on a predictive model despite the fact of including curated ODNs that have been well proven to be TLR-binding ligands.
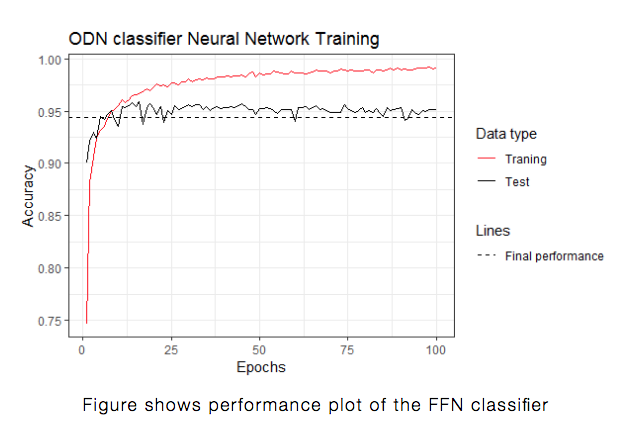
iGEMに出場したエジプトチームのウェブページに掲載されている図表を転載(http://2018.igem.org/Team:AFCM-Egypt/Machine_Learning)
合成生物の最適構成設計をAIを用いて探索する試みが広がると、AIの国際技術大会とiGEMの合同部会も生まれてくるかもしれない
AIの領域では、例えば、画像認識(一般物体認識)の精度を目指して、世界中の出場チームがAIアルゴリズムのパフォーマンスを競い合うILSVRCなどの国際競技会が、AIのそれぞれの領域別に、開かれています。
今後、合成生物の最適構成設計をAIを用いて探索する試みが広がるに従い、「合成生物の最適構成設計」のパフォーマンスを競い合うAIの国際技術大会が、iGEMとの合同部会として、誕生するのかもしれません。
[研究論文] 時系列深層学習モデル(LSTMほか)・畳込みニューラル・ネットワークモデルを用いた塩基配列解読・編集操作の結果予測モデル
なお、時系列深層学習モデル(LSTMほか)や畳込みニューラル・ネットワークモデルを用いて、塩基配列の解読したり、塩基配列の編集操作の結果を予測したりする試みに取り組んでいる様子を、数多くの論文に見ることができます。以下はその一部になります。
- Applied Bioinformatics
- DCNet — Denoising (DNA) Sequence With a LSTM-RNN and PyTorch
- Deep Learning in Bioinformatics
- Long short-term memory RNN for biomedical named entity recognition
- Improving protein disorder prediction by deep bidirectional long short-term memory recurrent neural networks.
- Deep Recurrent Neural Network for Protein Function Prediction from Sequence
- Convolutional LSTM Networks for Subcellular Localization of Proteins
- Comoputational modeling of cellular structures using conditional deep generative networks
- Multilayer encoder-decoder network for 3D nuclear segmentation in spheroid models of human mammary epithelial cell lines
- CNN を用いたバイオイメージのセグメンテーション
- バイオイメージ・インフォマティクス概要
- 全エクソンシーケンシングデータからの 生殖細胞系ゲノムコピー数予測ソフトウエアの調査
次回の記事で論じること
次回の記事では、研究論文ベースで、深層ニューラルネットワークモデルを含む機械学習・数理統計モデルが、遺伝子解読と遺伝子編集技術・遺伝子合成技術の精度の向上に対して、どのように貢献しているのか、その一旦を垣間見ていきたいと思います。